본 post는 LAIAD에서 제공하는 가톨릭의과대학 김태민 교수님의 Big Data in Precision Oncology를 정리한 내용입니다.
Intro
TCGA 멀티오믹스 데이터의 형태 및 분석 활용법을 학습합니다.
mRNA/miRNA의 형태 및 응용분석 예제를 학습합니다.
Mutation, SCNA, methylation의 형태 및 응용분석 예제를 학습합니다.
TCGA data 분석: mRNA/miRNA Expression
TCGA publication data에서 논문 출판에 사용된 RNA, RPPA, methylation 등 분석 data를 다운로드 받을 수 있습니다.
mRNA/miRNA expression data는 보통 아래와 같은 형태로 표현합니다. N개의 샘플별로 M개의 genes에 대한 각각 발현율을 숫자로 표시합니다. 이 때 발현율은 normalized intensity(microarray)나 RPKM/RSEM(RNA-seq)으로 표현합니다.
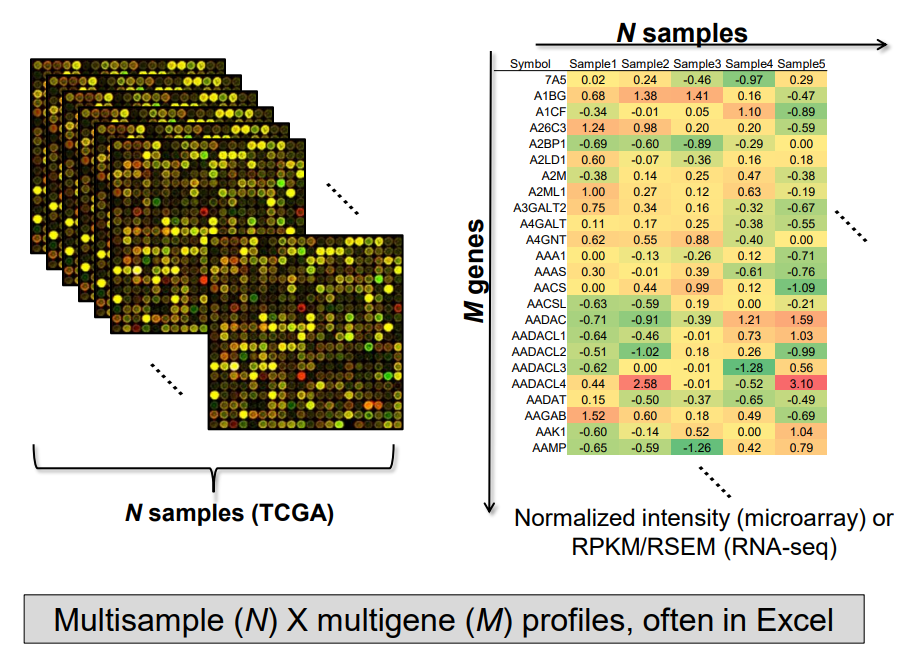 Standard Format for mRNA/miRNA Expression
Standard Format for mRNA/miRNA Expression
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
TCGA에서 mRNA/miRNA expression data를 다운로드 받아 살펴봅니다. 파일 size는 1.8G 정도로 큰 편에 속합니다. Standard format과 마찬가지로 각 샘플에 대해 gene별로 발현량이 표시되어 있습니다.
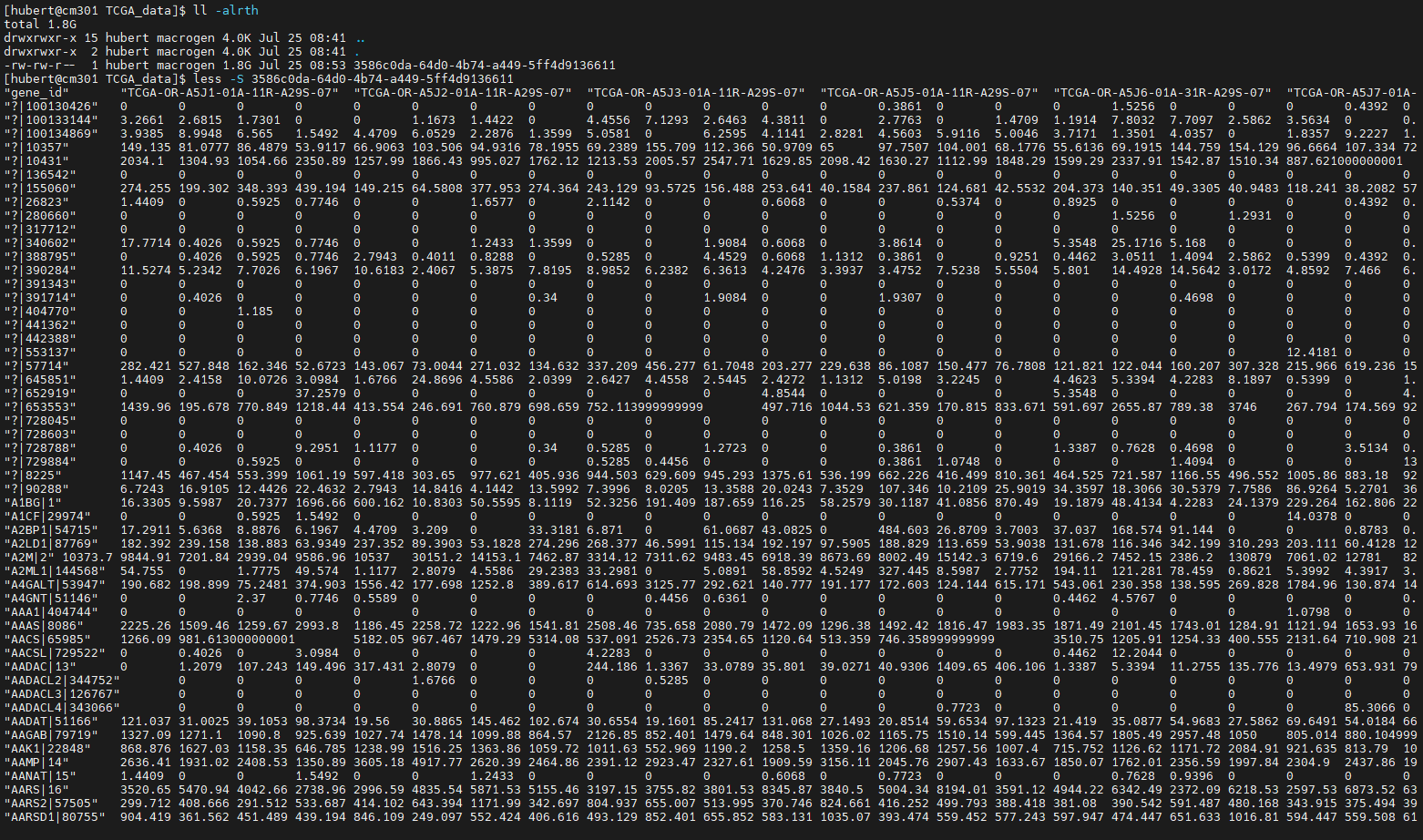 TCGA Format for mRNA/miRNA Expression
TCGA Format for mRNA/miRNA Expression
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
다운로드 받은 파일을 input으로 ‘consensus clustering’을 실습합니다.
1
2
3
4
5
6
7
8
data = read.table("unifiedScaledFiltered.txt", header=T, sep="\t", row.names=1)
head(data)
boxplot(data)
heatmap(as.matrix(data))
source("https://bioconductor.org/biocLite.R")
biocLite("ConsensusClusteringPlus") # install an R package!
library("ConsensusClusteringPlus") # load the installed package
result = ConsensusClusteringPlus(as.matrix(data), maxK=10, reps=1000, pItem=0.8, title="TCGA GBM", plot="pdf")
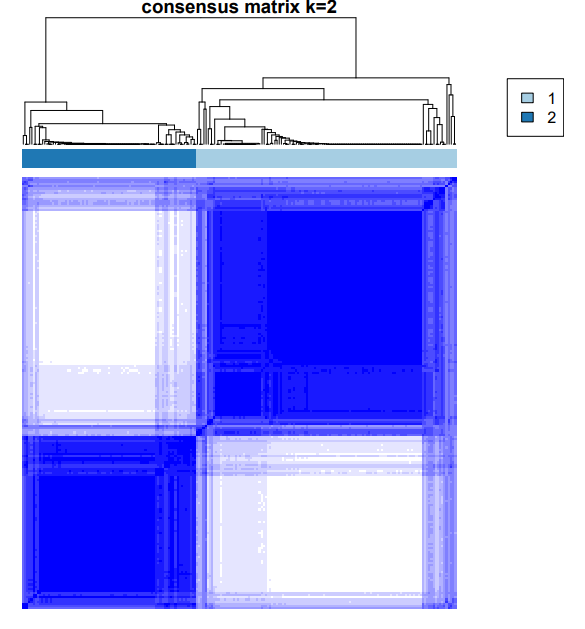 Consensus Clustering of GBM Expression Data
Consensus Clustering of GBM Expression Data
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
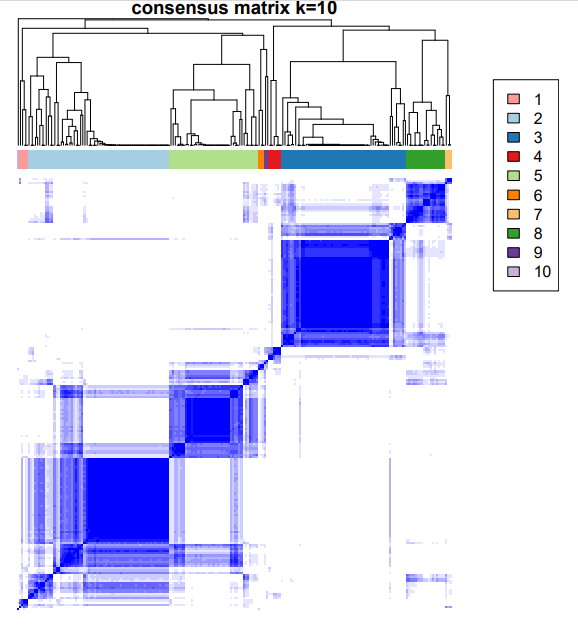 Consensus Clustering of GBM Expression Data
Consensus Clustering of GBM Expression Data
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
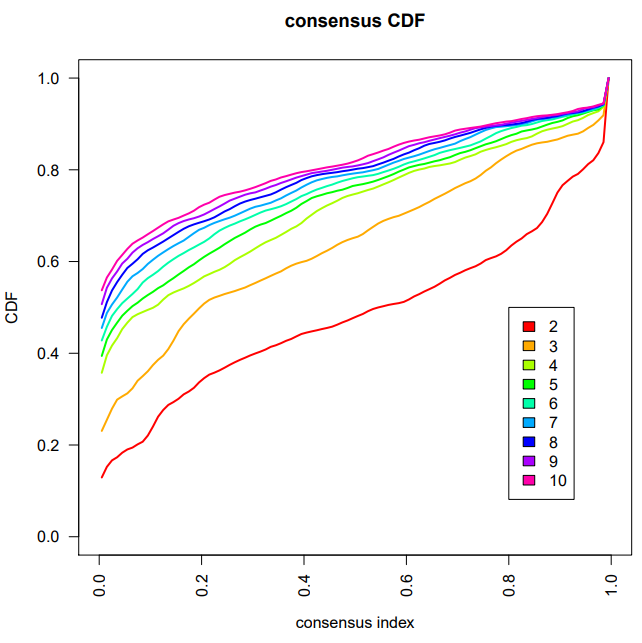 Consensus Clustering of GBM Expression Data
Consensus Clustering of GBM Expression Data
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
TCGA data 분석: DNA Copy Number Alterations
Copy number alterations는 microarray와 NGS 방식으로 모두 검출할 수 있습니다.
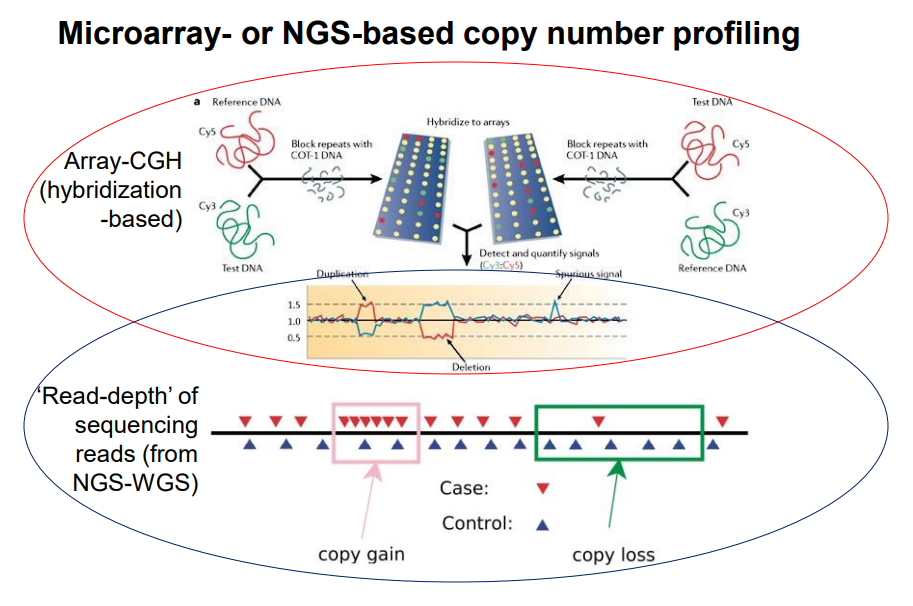 Copy Number Alterations
Copy Number Alterations
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
CNA data는 genome상의 각 구간별로 normalized ratio로 표현할 수 있습니다. 하지만 각 구간을 target으로 하는 probe 전체를 나열하면 굉장히 많은 양의 row가 필요합니다. 따라서 보통 비슷한 ratio를 가진 영역끼리 모아서 하나의 구간으로 표현하고 이를 seg 파일로 형태로 표현합니다. 아래 예시에서도 10개 구간으로 표현된 level2 data를 2개 구간으로 줄인 level3 data로 표현할 수 있습니다.
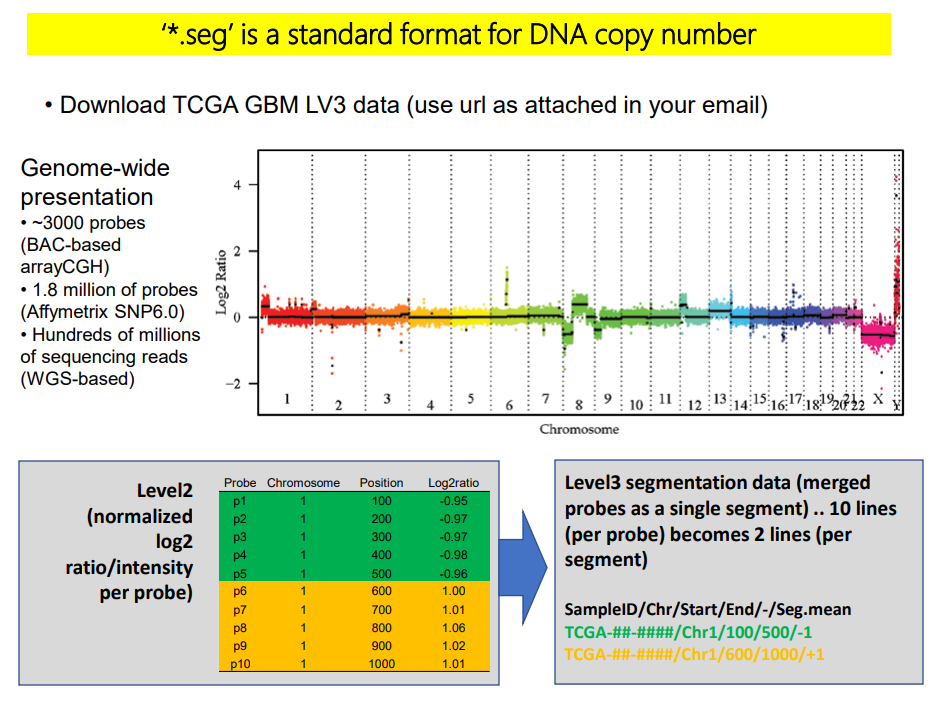 Seg 파일
Seg 파일
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
seg 파일은 IGV에 load해서 visualization 할 수 있습니다.
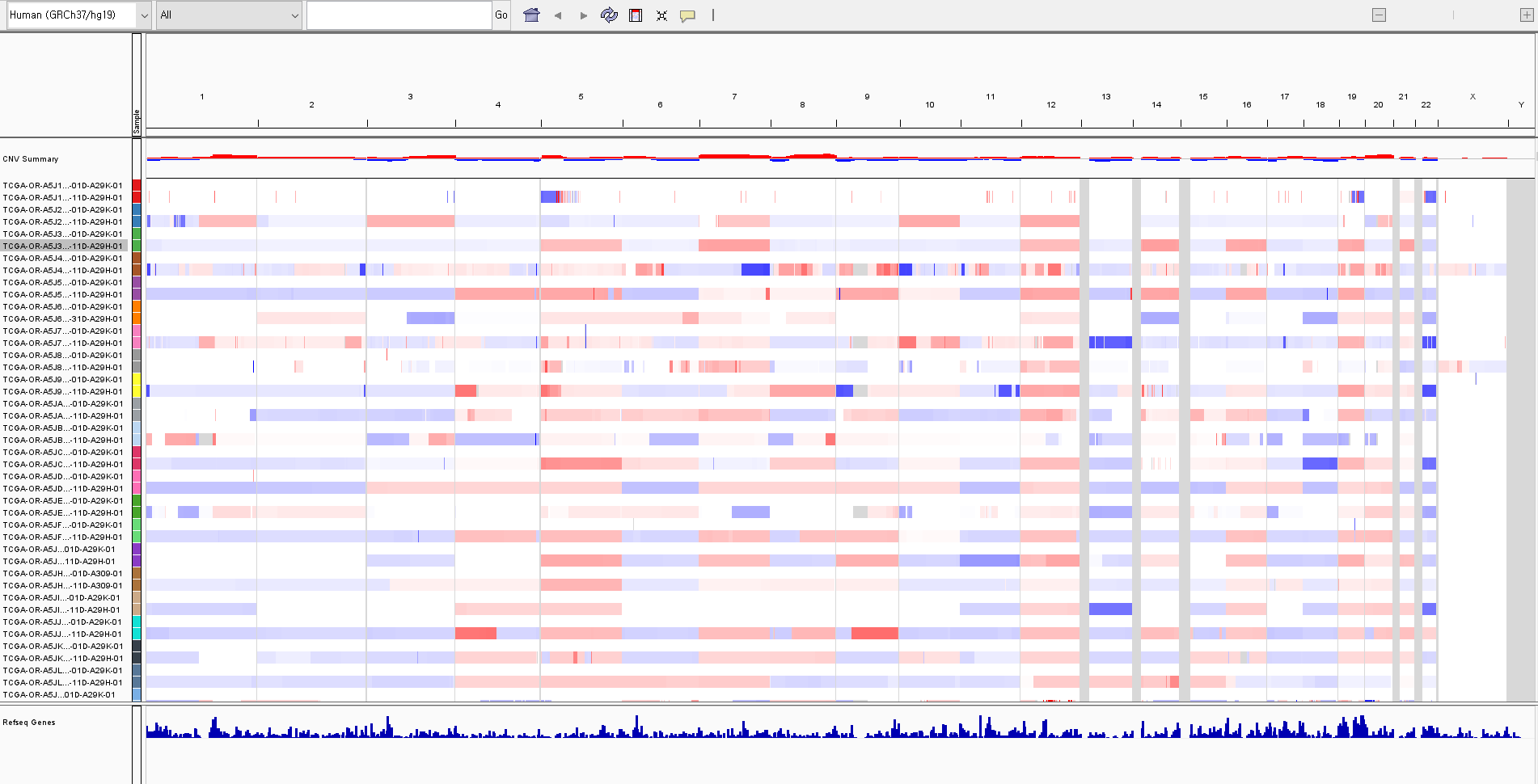 Seg on IGV
Seg on IGV
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
TCGA data 분석: Somatic Mutations
Mutation 분석 결과는 보통 vcf format으로 표기하며 가장 일반적인 format 입니다. TCGA는 방대한 project이므로 자체적으로 만든 mutation format을 사용했는데 이를 MAF(Mutation Annotation Format)라고 일컫습니다.
 VCF and MAF Format
VCF and MAF Format
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
Lung adenocarcinoma 환자가 가지고 있는 somatic mutation에 따라 EGFR inhibitor(Gefitinib)가 치료제로서 작동여부가 결정됩니다. Precision medicine의 대표적인 사례입니다.
 EGFR Inhibitor
EGFR Inhibitor
https://www.laidd.org/local/ubonline/view.php?id=141&group=1&returnurl=aHR0cHM6Ly93d3cubGFpZGQub3JnL2xvY2FsL3Vib25saW5lL2luZGV4LnBocD9vcmRlcnR5cGU9cmNfZCZrZXl3b3JkPUJJRyZwcm9ncmVzcyU1QiU1RD0xMyZlbnJvbF9zdGFydD0mZW5yb2xfZW5kPSZzdHVkeV9zdGFydD0mc3R1ZHlfZW5kPQ==
Take Home Message
- TCGA 멀티오믹스 데이터의 형태 및 분석 활용법에 대해 알아보았습니다.