본 post는 국가생명연구자원정보센터(KOBIC) 주관 서울대학교 박사 후 연구원 김준님의 유전체 지도 작성 개괄를 정리한 내용입니다.
Intro
유전체 지도 작성법에 대해 간략히 이해해 봅니다.
현존하는 세 가지 롱리드 시퀀싱 방식의 특징에 대해 알아봅니다.
2022년에 공개된 인간 유전체 지도 완성에 대해 알아봅니다.
유전체 지도와 유전체 정보
롱리드 시퀀싱은 한 번에 더 긴 길이의 리드를 생산해내며, 이들을 이어붙이는 과정(assembly)을 매우 쉽게 해줍니다.
유전체 지도는 이와 같이 리드들을 모아 이어붙여서 완성하게 됩니다.
유전체 지도 작성 관련 시퀀싱 플랫폼 특징
유전체 지도 작성 과정은 다음과 같습니다.
 유전체 지도 작성 과정
유전체 지도 작성 과정
https://www.edwith.org/longread-seq-2023/lecture/1475109
Genome assembly 관점에서 각 시퀀싱 데이터의 특징과 용도를 알아봅니다.
Arima Hi-C, Bionano optical map은 longread까지는 아니고 long range sequencing으로 분류됩니다.
PacBio HiFi가 가장 정확도가 높고 고품질의 시퀀싱 데이터를 얻을 수 있습니다. Size selection을 거치므로 10kb~20kb의 길이로 데이터를 생산합니다.
ONT ultra-long은 정확도가 떨어지지만 가장 긴 길이의 데이터를 생산합니다.
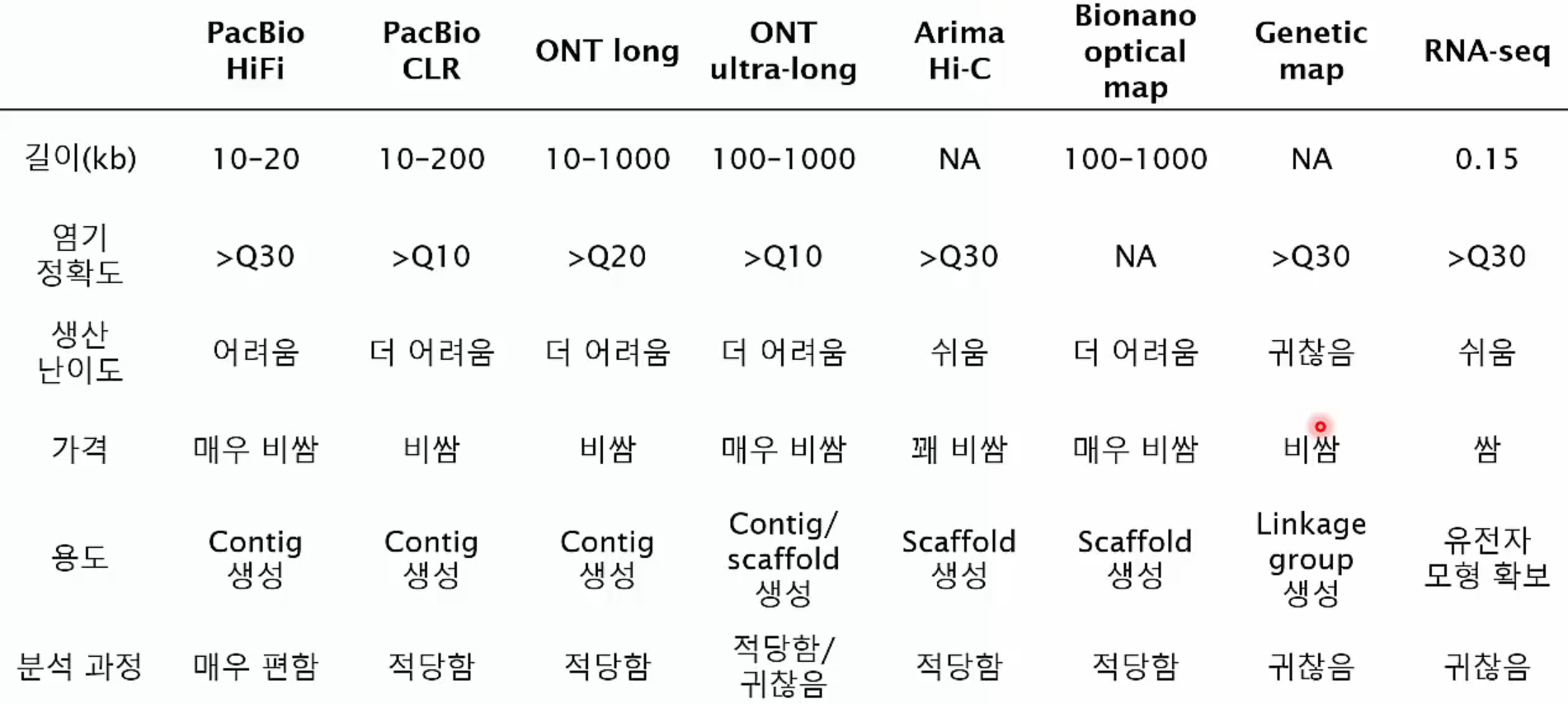 각 시퀀싱 데이터의 특징과 용도
각 시퀀싱 데이터의 특징과 용도
https://www.edwith.org/longread-seq-2023/lecture/1475109
유전체 지도를 생산하는 보편적인 과정은 다음과 같습니다.
Longread를 이어붙여 contig를 생산한 뒤, 이러한 contig들을 이어붙여 scaffold를 생성합니다. 이 때, physical map/optical map/genetic map 등을 활용하여 contig의 순서 및 방향을 결정합니다. 이후 RAN-seq을 수행하여 exon 정보 등을 확인합니다.
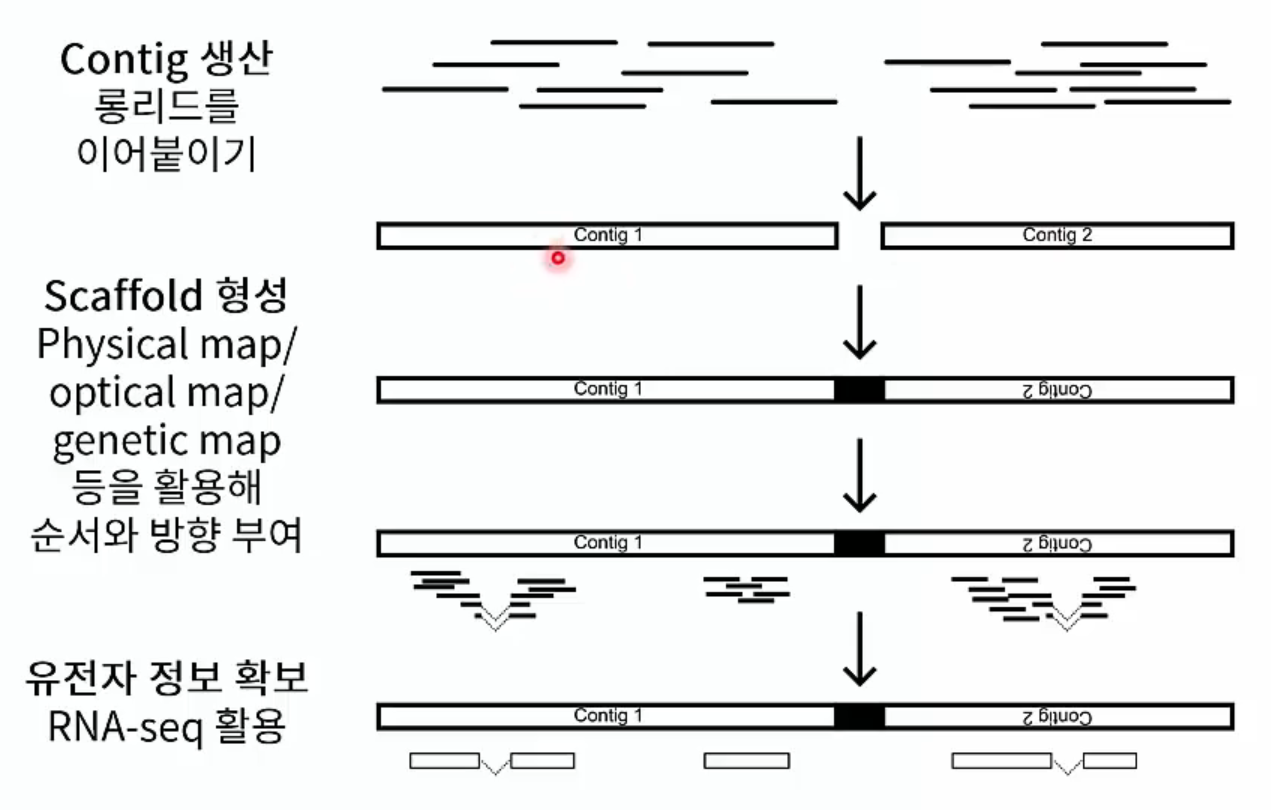 유전체 지도 생산 과정
유전체 지도 생산 과정
https://www.edwith.org/longread-seq-2023/lecture/1475109
유전체 지도를 작성할 때 assembly가 잘 안되는 지역이 존재합니다.
짧은 반복서열이 매우 많은 지역(repeat)
엄청 거대한 서열이 2회 이상 반복되는 지역(segmental duplication)
Heterozygosity가 문제될 정도로 큰 지역
centromere: 작은 subunit이 수 Mb까지 반복되기도 합니다.
Telomere & subtelomere: segmental duplication이 흔하게 관찰되는 지역입니다.
실제 사례
- CHM13 사용한 human genome
‘The complete sequence of a human genome’는 2022년 publish된, 현재 가장 완성에 가까운 human genome reference입니다. 해당 논문은 CHM13 cell line을 활용하여 유전체 지도의 완성도를 높혔습니다.
CHM은 완전포상기태(Complete Hydatidiform Mole, CHM) 세포주를 의미하며, 효과적으로 haploid cell line을 제작하는데 사용되고 있습니다. DNA가 없는 난자에 DNA가 있는 정자를 투입하면 정자 DNA만 2배로 불어난 세포가 되고 이것이 곧 homozygous cell line을 의미합니다. CHM13 cell line의 경우 세포 안에 존재하는 한 쌍의 염색체가 거의 동일하며, 이와 관련된 내용이 논문 내에 기재되어 있습니다.
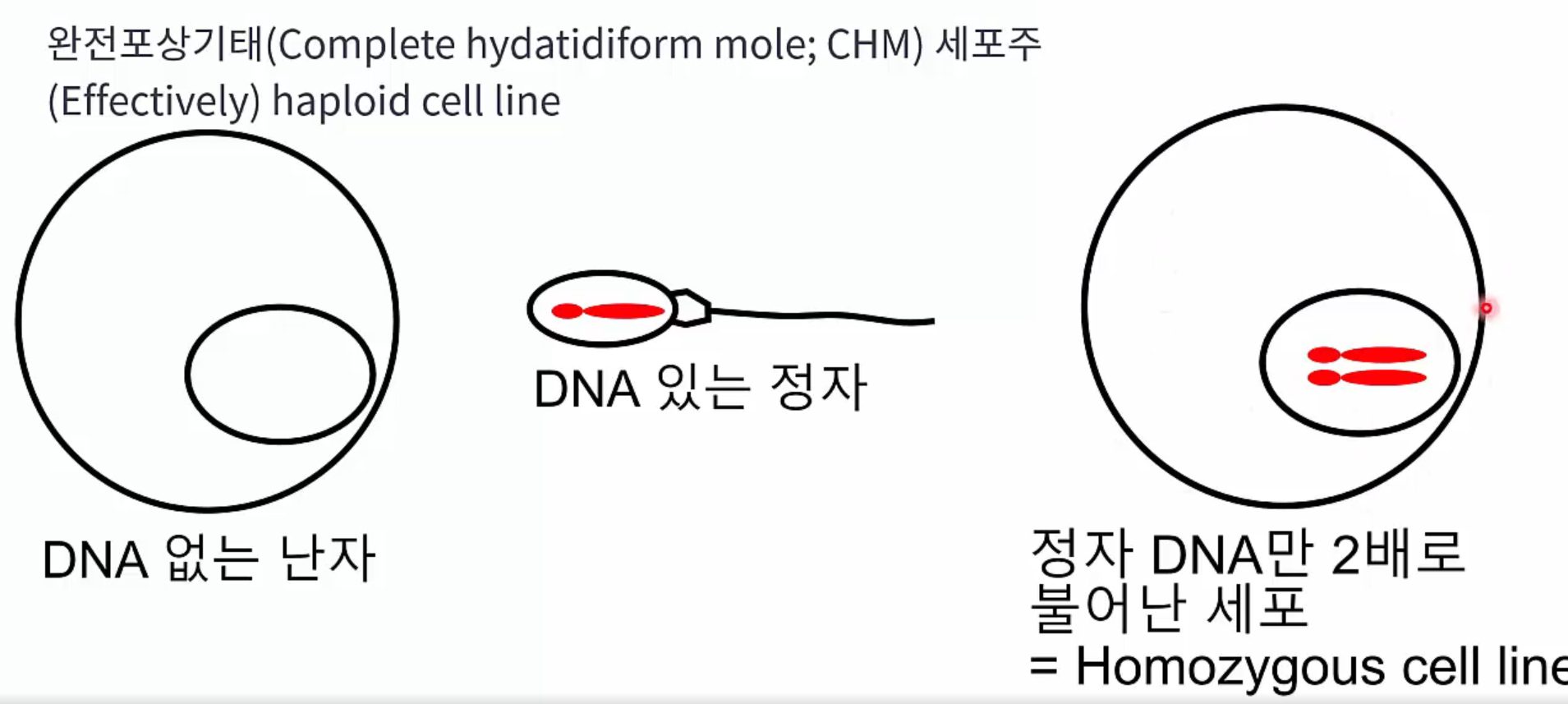 CHM13 Cell Line
CHM13 Cell Line
https://www.edwith.org/longread-seq-2023/lecture/1475109
해당 논문에서 기본 contig는 PacBio HiFi 리드를 사용하여 생산했습니다. 이를 사용하여 scaffold를 생산할 때 gap 영역은 N base로 채우는 것이 아니라 ONT ultra longread를 사용하여 시퀀스를 확인 했습니다.
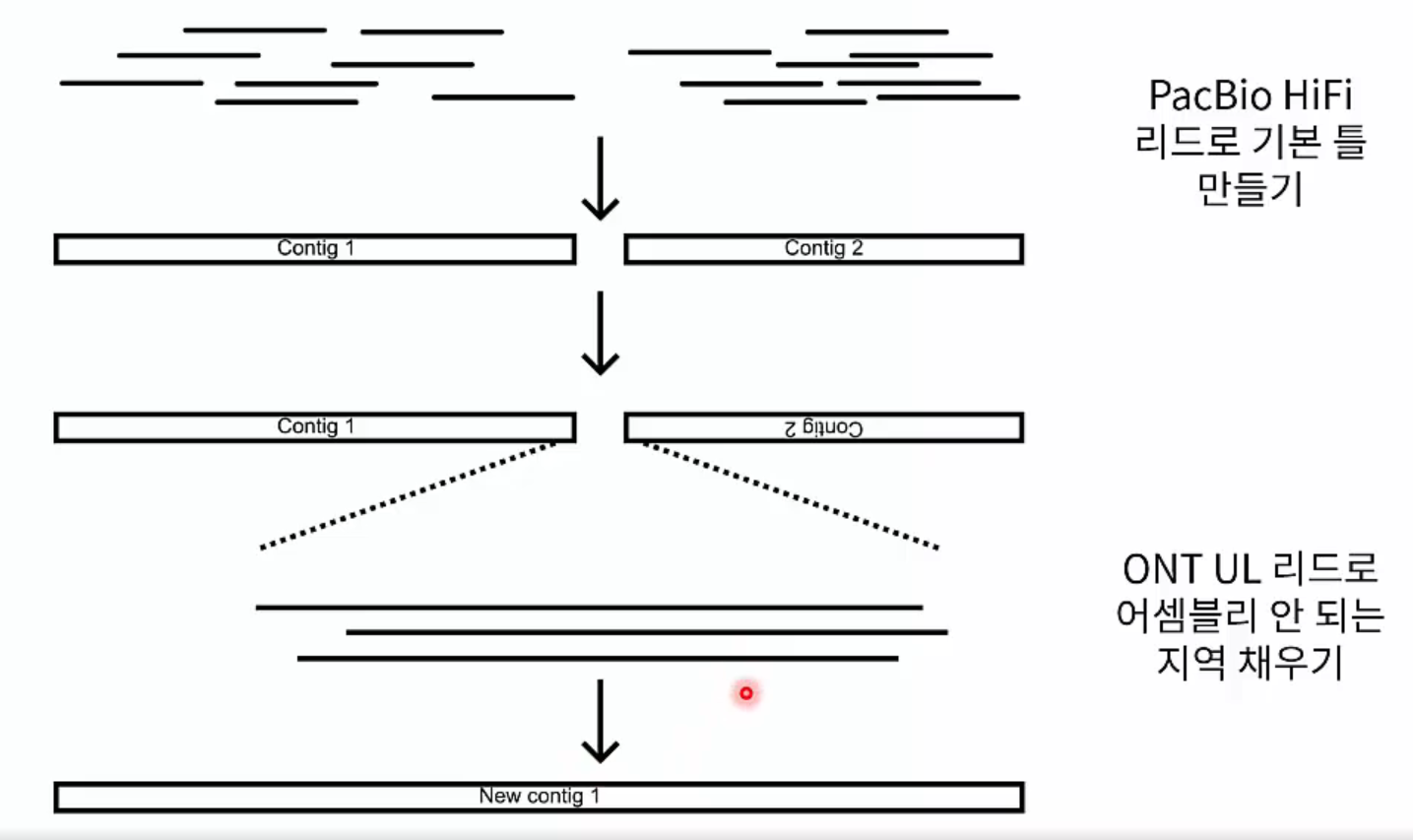 고품질 유전체 지도 생산 과정
고품질 유전체 지도 생산 과정
https://www.edwith.org/longread-seq-2023/lecture/1475109
- Human Pangenome Project
CHM cell line을 사용하여 유전체 지도를 만드는 과정에서 보다 scale up을 잘 할 수 있도록, 그리고 phasing을 잘되게 하는 것을 목적으로 하는 project입니다. Phasing은 모계에서 받은 DNA 정보, 부계에서 받은 DNA 정보를 나누는 과정입니다. PacBio HiFi + HiC + Strand-seq 등만 활용해도 diploid 개체의 유전체 정보 두 벌을 정확히 확인 가능합니다.
‘The Human Pangenome Project: a global resource to map genomic diversity’
‘A draft human pangenome reference’
Take Home Message
유전체 지도는 가장 고품질의 DNA 정보를 확보하는 과정입니다.
롱리드 사이에서 겹치는 부분을 활용해 contig를 만듭니다.
Physical/optical/henetic map 정보를 활용해 scaffold를 만듭니다.
RNA-seq 정보를 활용해 유전자 정보를 확보합니다.