본 post는 국가생명연구자원정보센터(KOBIC) 주관 서울대학교 박사 후 연구원 김준님의 유전체 크기 추정하기를 정리한 내용입니다.
Intro
유전체 크기를 추정하는 다양한 방법에 대해 알아봅니다.
시퀀싱 데이터로부터 직접 유전체 크기를 추정해봅니다.
앞으로 강의에서 다루는 분석내용은 STAR Protocols에 상세히 정리되어 있습니다.
유전체 크기 추정 - Wet 실험
유전체 크기를 추정해야 하는 이유는 다음과 같습니다.
생산해야 할 총 시퀀싱 양 & 시퀀싱 비용 계산에 필요합니다.
몇몇 genome assembler 작동시킬 때 option으로 넣어줘야 합니다.
사람 같이 이미 알려진 종에서는 생략 가능합니다.
유전체 크기 단위는 C-value를 사용합니다.
haploid DNA 질량 1pg은 대략 haploid genome size 1Gb와 유사하다고 볼 수 있습니다.
1pg(haploid DNA 질량) ~= 1Gb(haploid genome size)
실험적인 방법으로 genome size를 추정하는 방법은 다음과 같습니다.
- 형광 + flow cytometer
- 알려진 nucleus를 control로 삼고, 모르는 종의 nucleus 등을 뽑아 함께 염색합니다. 이후 형광 세기를 비교하여 genome size를 추정합니다.
- Single-copy gene + qPCR
- gDNA 추출 후 전체 질량 및 single-copy gene의 copy number를 측정합니다. 두 값을 활용하여 haploid genome size를 추정합니다.
- gDNA 추출 후 전체 질량 및 single-copy gene의 copy number를 측정합니다. 두 값을 활용하여 haploid genome size를 추정합니다.
유전체 크기 추정 - 알려진 DB
유전체 크기를 모아놓은 다양한 DB가 존재합니다.
- 약 6,222개 동물종에 대한 genome size data를 확인할 수 있습니다.
- 약 6,222개 동물종에 대한 genome size data를 확인할 수 있습니다.
- 약 12,273개 식물종에 대한 genome size data를 확인할 수 있습니다.
- 약 12,273개 식물종에 대한 genome size data를 확인할 수 있습니다.
유전체 크기 추정 - 시퀀싱 데이터
DNA 시퀀싱 기반 유전체 크기를 추정할 수 있습니다.
가상의 유전체 지도를 가정하고 생산한 DNA read응 유전체 지도에 mapping 합니다. 전체 생산 DNA 시퀀싱 총량을 depth로 나누어 유전체 크기를 추정할 수 있습니다.
예를들어, 5Gb의 시퀀싱 데이터를 생산하여 mapping 했을 때 약 5x depth로 mapping 되었다면, genome size는 약 1Gb라고 추정할 수 있습니다.
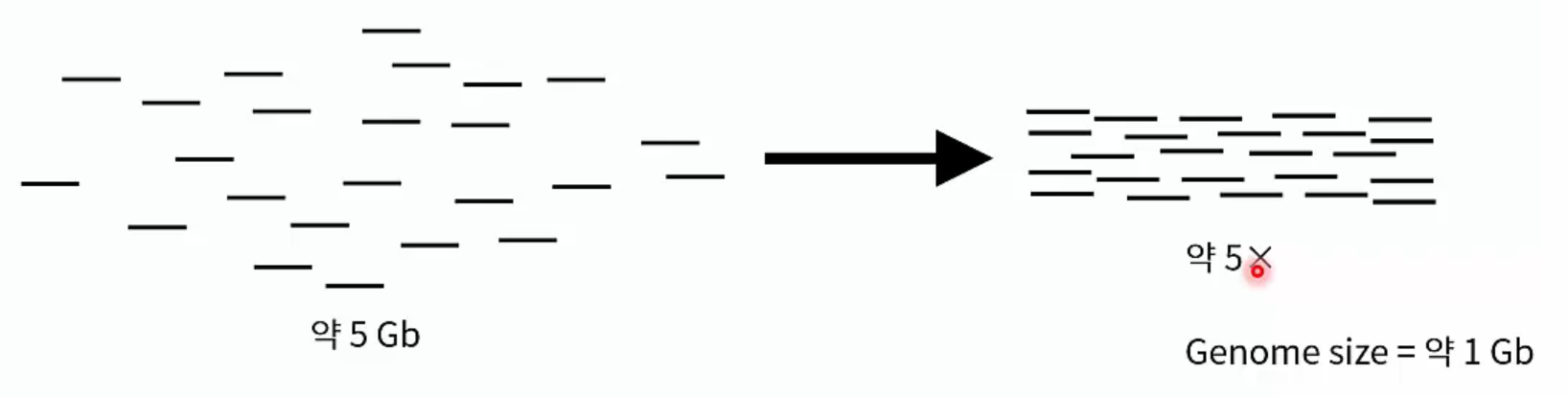 DNA 시퀀싱 활용한 유전체 크기 추정
DNA 시퀀싱 활용한 유전체 크기 추정
https://www.edwith.org/longread-seq-2023/lecture/1475110
DNA와 RNA 시퀀싱을 활용해서 유전체 크기를 추정할 수 있습니다.
Genome 대신 transcript 정보를 확보하고, 여기에 DNA read를 mapping해서 genome size를 추정합니다.
장점으로는 DNA data가 적어도 된다는 점입니다. 단점은 transcript quality가 좋지 않으면 genome size 추정이 부정확합니다.
 DNA+RNA 시퀀싱 활용한 유전체 크기 추정
DNA+RNA 시퀀싱 활용한 유전체 크기 추정
https://www.edwith.org/longread-seq-2023/lecture/1475110
Take Home Message
새로운 종의 genome assembly를 해야할 때는 유전체 크기 추정을 해야합니다.
다양한 방법이 있으며 시퀀싱 데이터만 가지고도 유전체 크기 추정이 가능합니다.