본 post는 국가생명연구자원정보센터(KOBIC) 주관 서울대학교 박사 후 연구원 김준님의 Repeat/Gene Annotation를 정리한 내용입니다.
Intro
유전자 정보를 확보하는 방법에 대해 알아봅니다.
반복서열을 masking하는 방법에 대해 알아봅니다.
Repeat Masking
Repeat masking을 하는 이유는 gene annotation을 좀 더 정확하고 선명하게 처리하기 위함입니다.
Gene annotation 과정을 살펴보면, 먼저 genome reference를 준비한 뒤 RNA-seq reads를 mapping합니다. 이후 gene annotation을 진행합니다. 이 때 repeat masking이 되어있으면 좀 더 정확하고 선명한 gene annotation 결과를 얻을 수 있습니다.
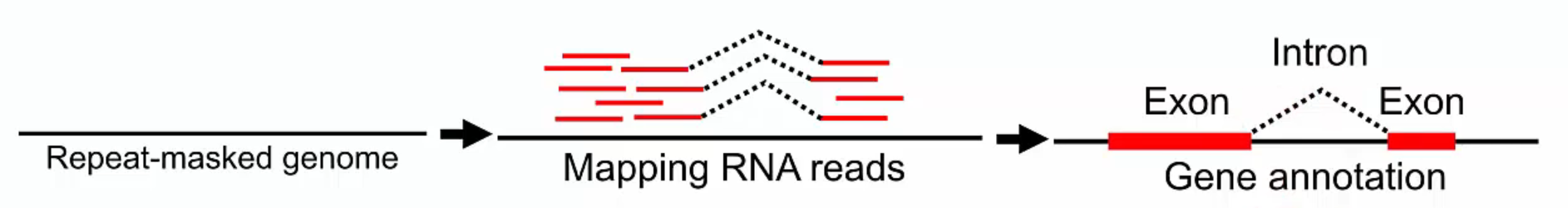 Gene Annotation Process
Gene Annotation Process
https://www.edwith.org/longread-seq-2023/lecture/1475112
다음은 Repeat 영역에 대해 알아보겠습니다.
많은 진핵생물의 유전체는 repeat으로 가득 차있습니다. HiFi를 비롯한 long-read sequencing 기법을 동원하면 repeat까지 정확하게 assemble 할 수 있습니다. 이런 repeat을 제거하거나 확인함으로써 유전체 활용을 더 효과적으로 할 수 있습니다.
Repeat masking은 두 가지 종류가 존재합니다.
Hard masking: repeat을 모두 N으로 masking하는 방법입니다.
Soft masking: repeat을 모두 소문자로 masking하는 방법입니다.
 Repeat Masking
Repeat Masking
https://www.edwith.org/longread-seq-2023/lecture/1475112
Gene Annotation
hisat2를 사용하여 masked genome에 RNA-seq data를 mapping합니다.
이후 braker, GeneMark를 다운로드 받아 설치한 뒤 실행합니다.
Take Home Message
Repeat masking -> RNA-seq read mapping -> gene annotation 순서로 진행합니다.