본 post는 국가생명연구자원정보센터(KOBIC) 주관 인사이트마이닝 이부일 CEO의 Analysis of variance with python을 정리한 내용입니다.
Intro
Python을 이용하여 Analysis of variance with python 수행과정을 알아봅니다.
언제 사용하는가?
Analysis of variance with python은 세 개 이상의 독립적인 모집단의 수치형 자료의 평균에 차이가 있는지 분석할 때 사용하는 방법입니다. 모든 모집단의 수치형 자료가 정규분포를 따를 때 사용할 수 있습니다.
가설 세우기
- 귀무가설 (Null Hypothesis, H0)
- 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 없습니다.
- 대립가설 (Alternative Hypothesis, H1 or HA)
- 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 있습니다.
- 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 있습니다.
 SST(Sum of Square Total)
SST(Sum of Square Total)
https://www.edwith.org/python-data-analysis-2023/lecture/1475049
- SST(Sum of Squuare Total)는 총제곱합으로 Y에 있는 모든 다름의 양을 의미합니다.
- SST = SSE + SSB
- SSE: 집단 내 제곱합을 의미합니다. 집단 내부적인 이유때문에 생긴 Y의 다름의 양입니다. 즉, SSE가 클수록 \(H_{0}\)(집단 간 차이가 없음)을 지지합니다.
- SSB: 집단 간 제곱합을 의미합니다. 집단이 달라서 생긴 Y의 다름의 양입니다. 즉, SSB가 클수록 \(H_{1}\)(집단 간 차이가 있음)을 지지합니다.
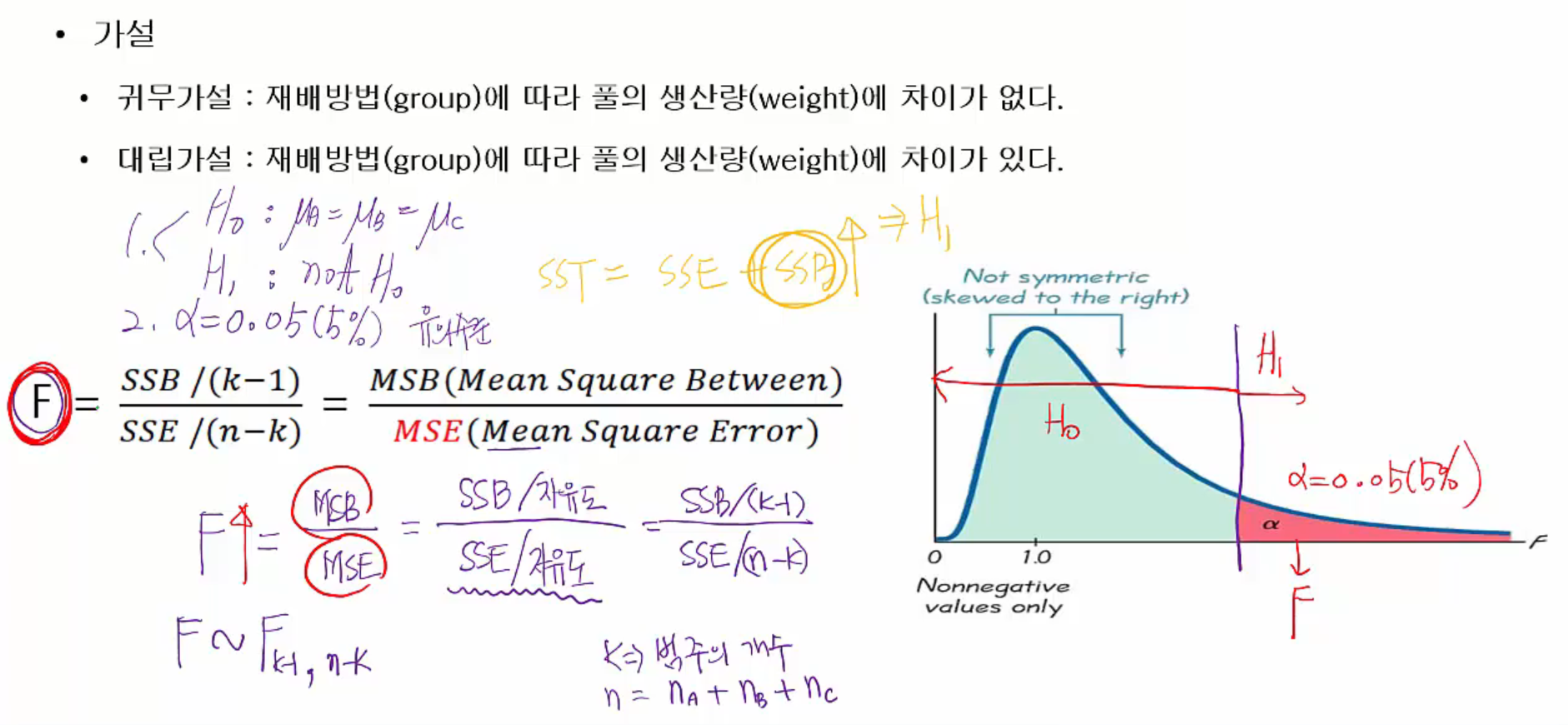 F분포
F분포
https://www.edwith.org/python-data-analysis-2023/lecture/1475049
Analysis of variance
- 1단계: 정규성 검정(Normality Test)
- 귀무가설: 정규분포를 따릅니다.
- 대립가설: 정규분포를 따르지 않습니다.
- n < 5,000: Shapiro-Wilk Normality Test
- n >= 5,000: Anderson-Darling Normality Test
- 정규성 검정은 control group에서 한 번, treat1 group에서 한 번, treat2 group에서 한 번, 총 세 번 시행합니다.
- 2단계: Levene의 등분산 검정
- 귀무가설: 등분산입니다.
- 대립가설: 이분산입니다.
- 3단계: 등분산이 가정된 ANOVA 또는 이분산이 가정된 ANOVA
- ANOVA를 시행합니다.
- ANOVA를 시행합니다.
실습
- 가설 설정
- 귀무가설: 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 없습니다.
- 대립가설: 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 있습니다.
 데이터 로딩
데이터 로딩
https://www.edwith.org/python-data-analysis-2023/lecture/1475049
- 분석 1단계: 정규성 검정(Normality Test)
- 귀무가설: 정규분포를 따릅니다.
- 대립가설: 정규분포를 따르지 않습니다.
- n < 5,000 : Shapito-Wilk Normality Test (shapiro(data.variable))
- n >= 5,000 : Anderson-Darling Normality Test (anderson(data.variable))
- shapiro normality test 결과,
- ctrl group: 유의확률(p-value)가 0.747로 정규성 가정을 만족합니다.
- trt1 group: 유의확률(p-value)가 0.452로 정규성 가정을 만족합니다.
- trt2 group: 유의확률(p-value)가 0.564로 정규성 가정을 만족합니다.
 정규성 검정
정규성 검정
https://www.edwith.org/python-data-analysis-2023/lecture/1475049
- 분석 2단계: Levene의 등분산 검정
- 귀무가설: 등분산입니다.
- 대립가설: 이분산입니다.
- levene 등분산 검정 결과 유의확률(p-value) 0.341로 유의수준(0.05)보다 크므로 귀무가설을 기각하지 못합니다. 즉, 등분산입니다.
 Levene의 등분산 검정
Levene의 등분산 검정
https://www.edwith.org/python-data-analysis-2023/lecture/1475049
- 분석 3단계: Analysis of variance
- 귀무가설: 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 없습니다.
- 대립가설: 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 있습니다.
- 유의확률(p-value) 0.016으로 유의수준(0.05)보다 작으므로 귀무가설을 기각합니다. 즉, 재배방법(group)에 따라 풀의 생산량(weight)에 차이가 있습니다.
 ANOVA
ANOVA
https://www.edwith.org/python-data-analysis-2023/lecture/1475049
코드
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
import pandas as pd
import scipy.stats as stats
PlantGrowth = pd.read_excel(
io = '07PlantGrowth.xlsx',
sheet_name = 0
)
print(PlantGrowth)
# Analysis I: Normality Test
# n < 5,000 -> Shapiro-Wilk Normality Test
print(stats.shapiro(PlantGrowth.loc[PlantGrowth['group'] == 'ctrl', 'weight']))
print(stats.shapiro(PlantGrowth.loc[PlantGrowth['group'] == 'trt1', 'weight']))
print(stats.shapiro(PlantGrowth.loc[PlantGrowth['group'] == 'trt2', 'weight']))
# Analysis II: Levene의 등분산 검정
# 귀무가설: 등분산입니다.
# 대립가설: 이분산입니다.
stats.levene(
PlantGrowth.loc[PlantGrowth['group'] == 'ctrl', 'weight'],
PlantGrowth.loc[PlantGrowth['group'] == 'trt1', 'weight'],
PlantGrowth.loc[PlantGrowth['group'] == 'trt2', 'weight'],
)
# Analysis III: ANOVA
stats.f_oneway(
PlantGrowth.loc[PlantGrowth['group'] == 'ctrl', 'weight'],
PlantGrowth.loc[PlantGrowth['group'] == 'trt1', 'weight'],
PlantGrowth.loc[PlantGrowth['group'] == 'trt2', 'weight'],
)
Take Home Message
Analysis of variance 이론을 학습하고 Google colab에서 실습해 보았습니다.