Sequencing 개요
about DNA post에서 DNA의 화학구조와 특성을 확인 했습니다. Sequencing의 목적은 pentose sugar의 1’ carbon에 연결된 nitrogenous base의 순서를 확인하는 것임을 또한 확인 했습니다.
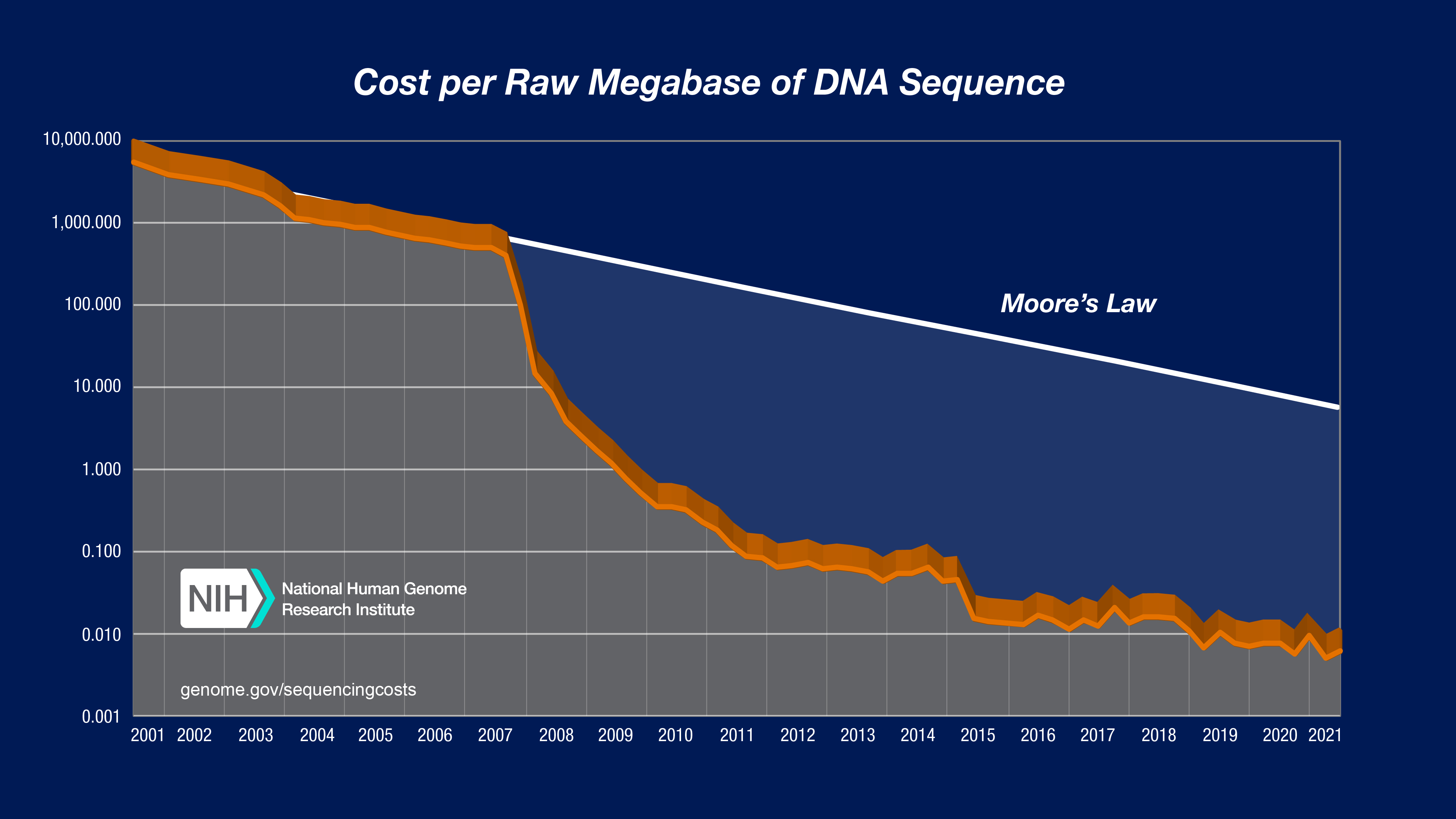 Sequencing cost per megabases - 2021
Sequencing cost per megabases - 2021
https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Costs-Data
 Sequencing cost per Human Genome data - 2021
Sequencing cost per Human Genome data - 2021
https://www.genome.gov/about-genomics/fact-sheets/DNA-Sequencing-Costs-Data
Technology는 단점을 보완하여 항상 발전합니다. Sequencing tech는 반도체 못지 않게 급격한 발전을 이룬 분야 중 하나입니다. 위 그림은 sequencing tech와 관련된 강의, 세미나의 첫 시간에 자주 언급되는 내용입니다. (마크로젠 PT 면접에서도 위 그림을 포함 했…) “Moore's Law”는 인텔의 Gordon Moore가 1965년 발표한 법칙으로, 반도체에 들어가는 transistor의 수가 24개월마다 2배 증가한다는 것입니다. 이와 비교해서 megabase당 sequencing 비용과 Human Genome seqeuncing 비용은 더 급격히 감소하고 있습니다. Y-axis가 log scale임을 감안할 때 가격의 하락폭은 훨씬 더 급격하다는 것을 알 수 있습니다. 대용량 sequencing capability를 가진 Illumina의 Novaseq이 2017년 출시하면서 비로소 1,000 dollars Human Genome era를 맞이하게 되었습니다. 1990년부터 2003년까지 진행된 The Human Genome Project가 13년의 시간, $3 bilion dollars(3조원)이 소요된 것과 비교하면 가히 놀라울 만한 발전이 아닐 수 없습니다.
1st Generation Sequencing: Sanger Sequencing
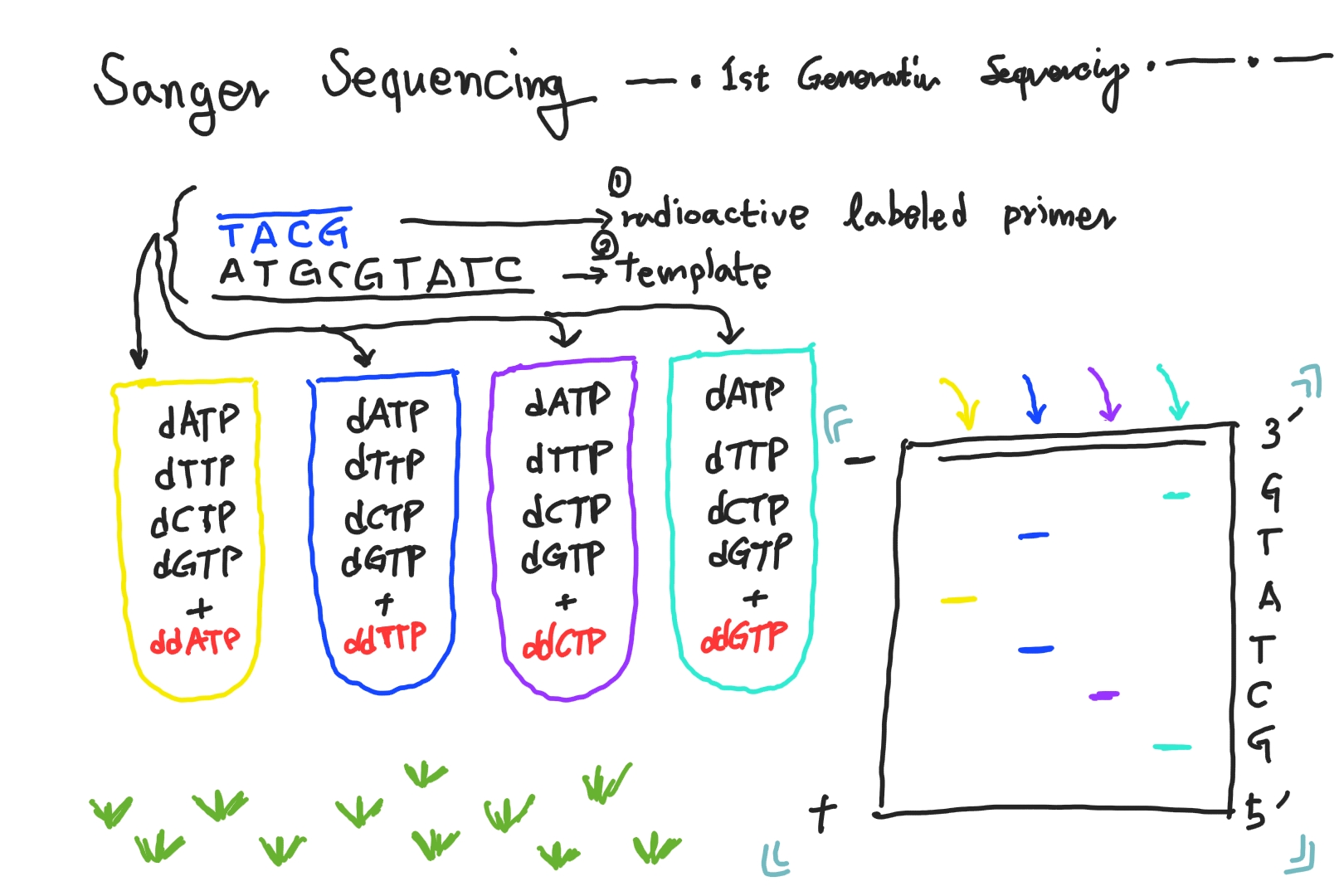 Sanger Sequencing
Sanger Sequencing
Sanger sequencing은 1977년 Frederick Sanger와 동료들에 의해 개발된 sequencing 입니다. ddNTP(Dideoxynucleotide TriPhosphate)의 선택적 결합에 의해 DNA 복제가 종료되는 원리를 사용하여 base sequence를 확인합니다.
우선 baseq sequence를 알고자 하는 template(single strand) DNA를 준비합니다. DNA elongation에 필요한 DNA primer도 준비합니다. DNA polymerase와 함께 dNTP, ddNTP가 담긴 tube에 준비물들을 넣으면 랜덤하게 길이가 다른 DNA fragment가 생성됩니다. dNTP가 결합하면 elongation이 계속 진행되지만, ddNTP가 결합하면 elongation을 이어갈 hydroxyl group(-OH)이 없으므로 반응은 종결됩니다. 마지막으로 gel electrophoresis(전기영동)로 DNA fragment를 내려주면 길이에 따라 band가 뜨고 base sequence를 확인할 수 있습니다.
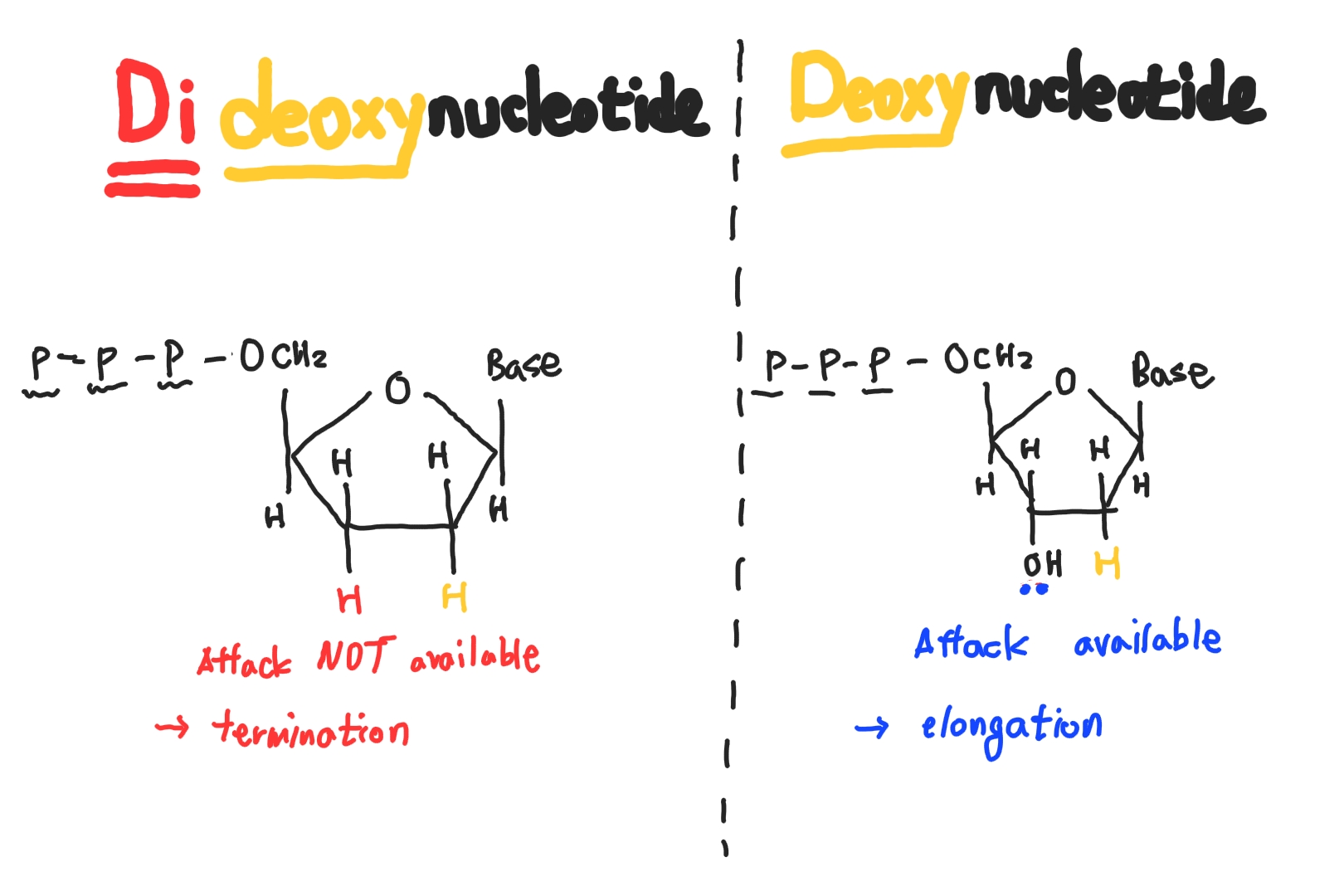 Dideoxynucleotide TriPhosphate
Dideoxynucleotide TriPhosphate
dNTP(Deoxynucleotide TriPhosphate)와 달리 ddNTP(Dideoxynucleotide TriPhosphate)는 3’ -H group만 있으므로 더이상 새로운 nucleotide와 결합하지 못하고 elongation 반응이 종결됩니다.
Sanger sequencing은 확인하고자 하는 부위를 PCR 증폭해야 하고, 한 번 반응할 때 확인 가능한 base 길이에 한계가 있습니다. 이런 단점을 극복하기 위해 NGS가 개발되었으며, 그 전까지 약 25년 동안 가장 널리 사용된 DNA sequencing 기술입니다.
오늘날 업계에서는 여전히 sanger sequencing을 사용합니다. 다만 전통적인 sanger sequencing에서 좀 더 개선된 automated sanger sequencing을 사용합니다. 원리는 똑같으나 base sequence 확인 과정을 자동화하여 소요 시간을 단축시킨 방식입니다. 주로 짧은 길이의 base seqeuence를 정확하게 확인하거나 NGS 결과를 검증할 목적으로 sanger sequencing을 사용합니다.
2nd Generation Sequencing: NGS
NGS는 cloning이 필요없고 library fragment의 amplification과 sequencing이 동시다발적으로 진행되기 때문에 비용과 소요시간을 절감할 수 있습니다. 2세대 sequencing이라고도 불리는 NGS는 short DNA 가닥을 만들어 squencing 하는 것이 특징이며, Illumina의 HiSeq, NovaSeq과 Thermo Fisher Scientific의 Ion Torrent가 대표적인 기기입니다. 2010년 이후 비약적인 발전을 이루어 왔습니다.
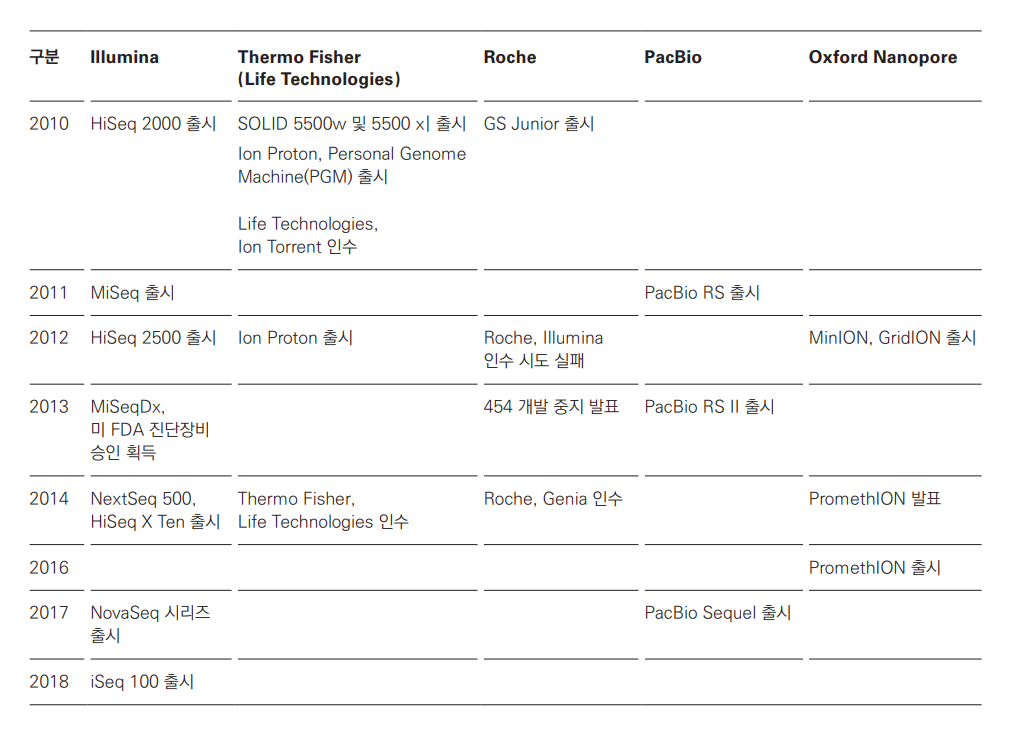 2010년 이후 주요 NGS 회사의 플랫폼 변화
2010년 이후 주요 NGS 회사의 플랫폼 변화
‘Next Generation Sequencing 기반 유전자 검사의 이해(2019) -식품의약품안전처-
NGS는 clonal template 생성 방식에 따라 SBL(sequencing by ligation)과 SBS(sequencing by synthesis)로 나눌 수 있습니다.
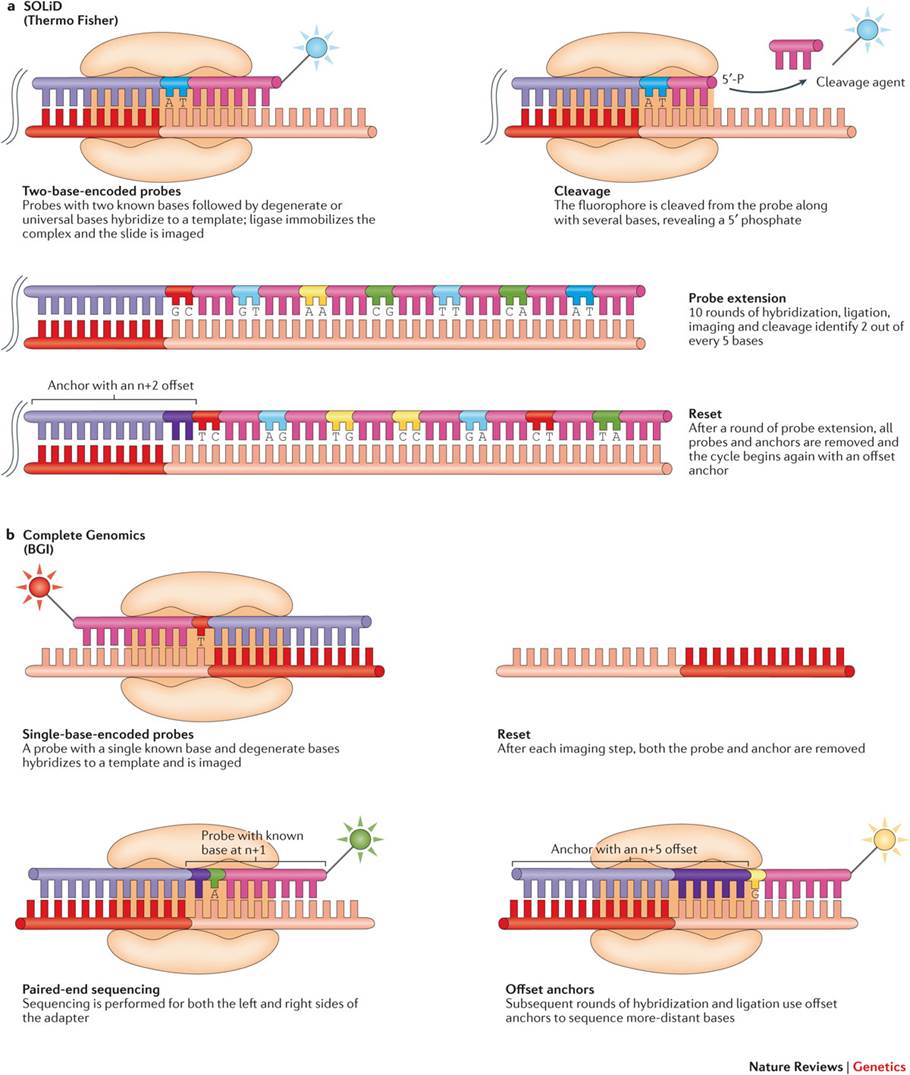
SBL(sequencing by ligation)
Ligase와 Fluorescentyl labeled probe를 사용하여 sequence를 알아내는 방식입니다. 1~2bp 길이의 known base로 구성된 fluorescently labeled probe가 target DNA fragment에 결합한 뒤 인접한 2~10bp 길이의 oligonucleotide가 연결됩니다. 이 결합으로 인해 fluorescently labeled probe가 특정 spectrum의 빛을 방출하는데 이를 분석하여 base를 확인합니다.
Life Technologies의 SOLiD(Sequencing by Oligonucleotide Ligation and Detection)와 Complete Genomics의 cPAL(combinatorial Probe-Anchor Ligation)이 SBL 방식의 대표적인 사례입니다.
![Post-Image]() Sequencing by ligation methods
Sequencing by ligation methods
Goodwin, S., McPherson, J. & McCombie, W. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet 17, 333–351 (2016). https://doi.org/10.1038/nrg.2016.49SBS(sequencing by synthesis)
DNA polymerase와 Fluorescentyl labeled base를 사용하여 sequence를 알아내는 방식입니다. SBS는 다시 CRT(cyclic reversible termination)와 SNA(single-nucleotide addition)로 나눌 수 있습니다.
CRT(cyclic reversible termination)
Sanger sequencing과 마찬가지로 3’-OH group이 막힌 deoxyribose를 사용합니다. DNA template의 adapter region에 primer가 결합한 뒤 polymerase가 달라붙습니다. 네 가지 종류의 dNTPs(deoxynucleotides)가 투입되고 그 중 DNA template에 상보적인 한 개의 dNTP가 결합합니다. 결합하지 못한 나머지 dNTPs는 wash-out 시킵니다. 각각의 cluster에 결합한 한 개의 dNTP에 붙어있던 flurophore가 떨어지면서 빛을 방출하고 이를 분석하여 sequence를 확인합니다.
Illumina와 Qiagen의 sequencer가 CRT 방식의 대표적인 사례입니다.
![Post-Image]() Sequencing by synthesis: cycliv reversible termination approaches
Sequencing by synthesis: cycliv reversible termination approaches
Goodwin, S., McPherson, J. & McCombie, W. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet 17, 333–351 (2016). https://doi.org/10.1038/nrg.2016.49SNA(single-nucleotide addition)
CRT와 달리 한 번에 한 개의 dNTP를 투입하여 a single signal을 확인하는 방식입니다.
454 Life Sciences의 pyrosequencing과 Thermo Fisher Scientific의 Ion Torrent가 SNA 방식의 대표적인 사례입니다.
![Post-Image]() Sequencing by synthesis: single-nucleotide addition approaches
Sequencing by synthesis: single-nucleotide addition approaches
Goodwin, S., McPherson, J. & McCombie, W. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet 17, 333–351 (2016). https://doi.org/10.1038/nrg.2016.49
3rd Generation Sequencing: Long-read Sequencing
2nd generation sequencing의 단점인 short-read를 보완한 sequencer로 PCR amplification 없이 DNA strand의 real-time sequencing 방식을 통해 long-read를 한 번에 읽을 수 있습니다. Single base quality가 높지 않아서 한계가 있지만 점차 정확도를 높히면서 임상진단 분야로 확대하고 있습니다.
크게 두 가지 종류로 나눌 수 있는데, SMRT(single-molecule real-time) sequencing과 synthetic 방식입니다.
SMRT(Single-molecule long-read) sequencing
현재 SMRT를 널리 사용하는 회사는 Pacific Biosciences입니다. SMRT는 특별한 flow cell을 사용하는데, ZMW(zero-mode waveguides)로 불리는 transparent bottoms를 지닌 picolitre 단위의 수 천 개 well이 존재합니다. Short-read SBS technology는 polymerase가 DNA를 따라서 움직이며 elongation이 진행되는 반면, PacBio는 DNA polymerase가 well 바닥에 고정되어 있고 DNA strand가 ZMW를 통과하며 sequence를 확인하는 방식입니다. Well은 단 한 개의 DNA strand만 통과 가능하며, dNTP가 결합하면서 방출하는 빛을 laser와 camera가 계속 확인합니다. 길이가 3kb 이상인 DNA는 어렵지만 그 이하의 DNA fragment는 여러 차례 반복해서 sequencing 될 수 있으며 CCS(circular consensus seqeunce)로 알려진 consensus read of insert를 생성합니다.
2014년 Oxford Nanopore Technologies(ONT)에서 MinION을 발표했습니다. 다른 회사의 sequencer들은 빛, 색상, pH와 같은 secondary signal을 사용한 반면, nanopore는 naive ssDNA의 sequence를 직접 읽습니다.
Synthetic long-reads
그동안 확인했던 다른 sequencing platforms와 달리 short-read seqeuncer로 생산한 read와 각 fragment에 부여된 barcode에 의존하여 synthetic long-read를 구현하는 방식입니다.
Illumina의 synthetic long-read sequencing과 10X Genomics의 emulsio-based system이 대표적인 사례입니다.
![Post-Image]() Sequencing by synthesis: single-nucleotide addition approaches
Sequencing by synthesis: single-nucleotide addition approaches
Goodwin, S., McPherson, J. & McCombie, W. Coming of age: ten years of next-generation sequencing technologies. Nat Rev Genet 17, 333–351 (2016). https://doi.org/10.1038/nrg.2016.49
NGS 장비의 장단점
NGS sequencer마다 원리가 다르기 때문에 서로 다른 장단점을 가지고 있습니다. Illumina, Thermo Fisher Scientific으로 대표되는 2nd generation sequencing은 read length가 짧아서 정확도가 높고 SNP 검출에 유리하지만, structural variants 검출에는 취약합니다.
반면 PacBio, Oxford Nanopore Technologies로 대표되는 3rd generation sequencing은 long-read의 이점을 살려 de-novo assembly와 structural variants 검출에 유리하지만, 정확도가 낮고 SNV 검출에 취약합니다.
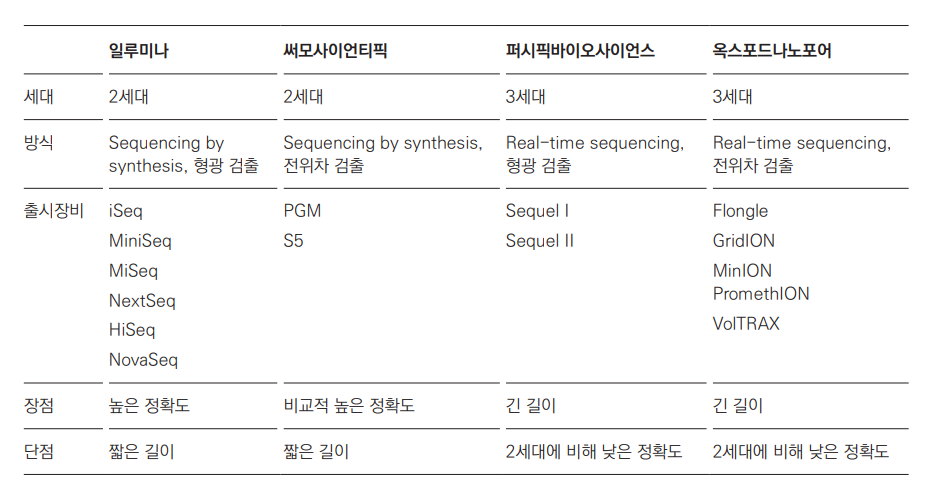 NGS sequencer의 장단점
NGS sequencer의 장단점
‘Next Generation Sequencing 기반 유전자 검사의 이해(2019) -식품의약품안전처-
Take Home Message
DNA와 RNA의 구조는 molecular biology와 organic chemistry 수업 시간에 배운 기억이 있습니다. 당시에는 외워야하는 딱딱한 지식으로 생각했지만 DNA, RNA에서 일어나는 모든 화학반응과 NGS, bioinformatics 목적을 이해하기 위해서는 기본 구조를 잘 알아야 한다는 것을 다시 한 번 느꼈습니다. 또한 DNA replication과 protein 발현까지 진행되는 central dogma 원리를 알아야 중간에 문제가 발생해서 질병으로 이어지는 현상도 이해할 수 있습니다.
Sequencing technology는 발전속도가 굉장히 빠른 분야입니다. 머지 않아 ssDNA를 짧은 시간 안에 저렴한 비용으로 sequencing 할 수 있는 시대가 찾아오지 않을까요?
 Sequencing by ligation methods
Sequencing by ligation methods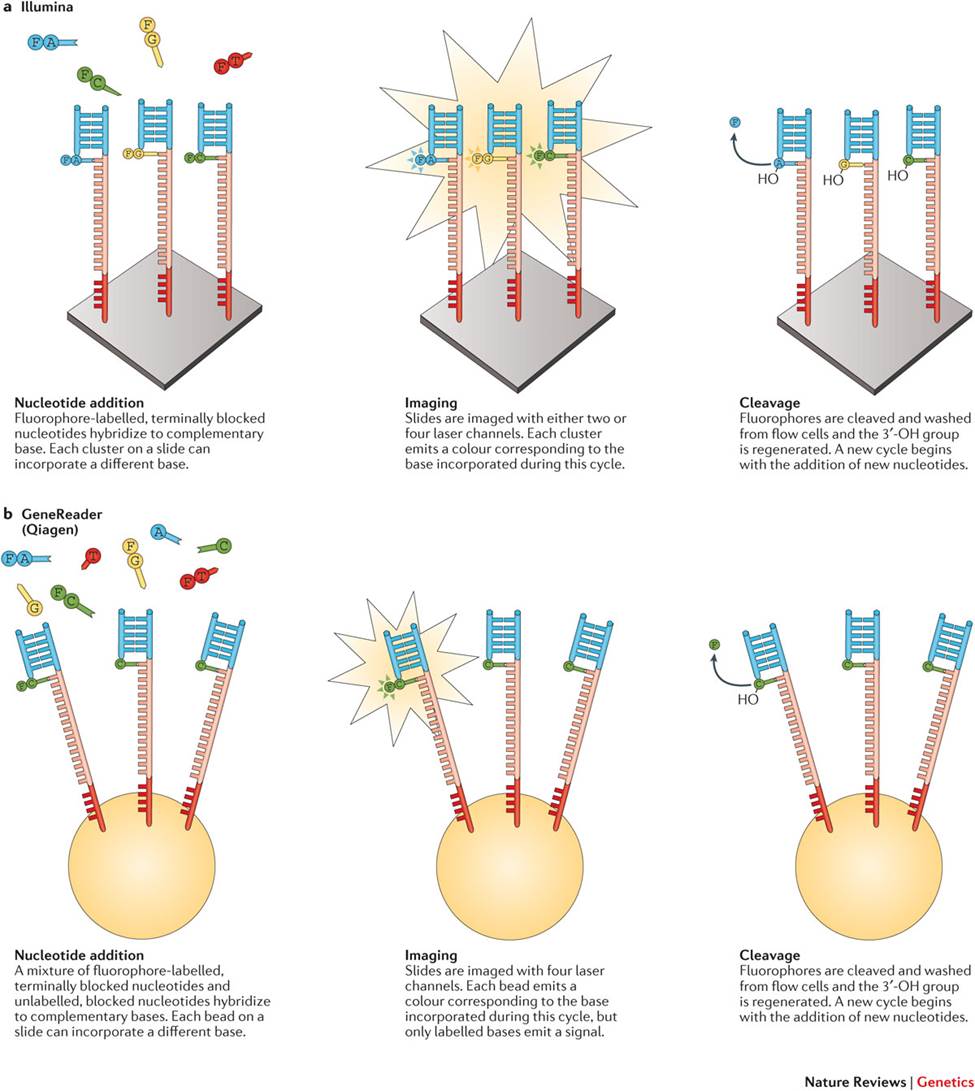 Sequencing by synthesis: cycliv reversible termination approaches
Sequencing by synthesis: cycliv reversible termination approaches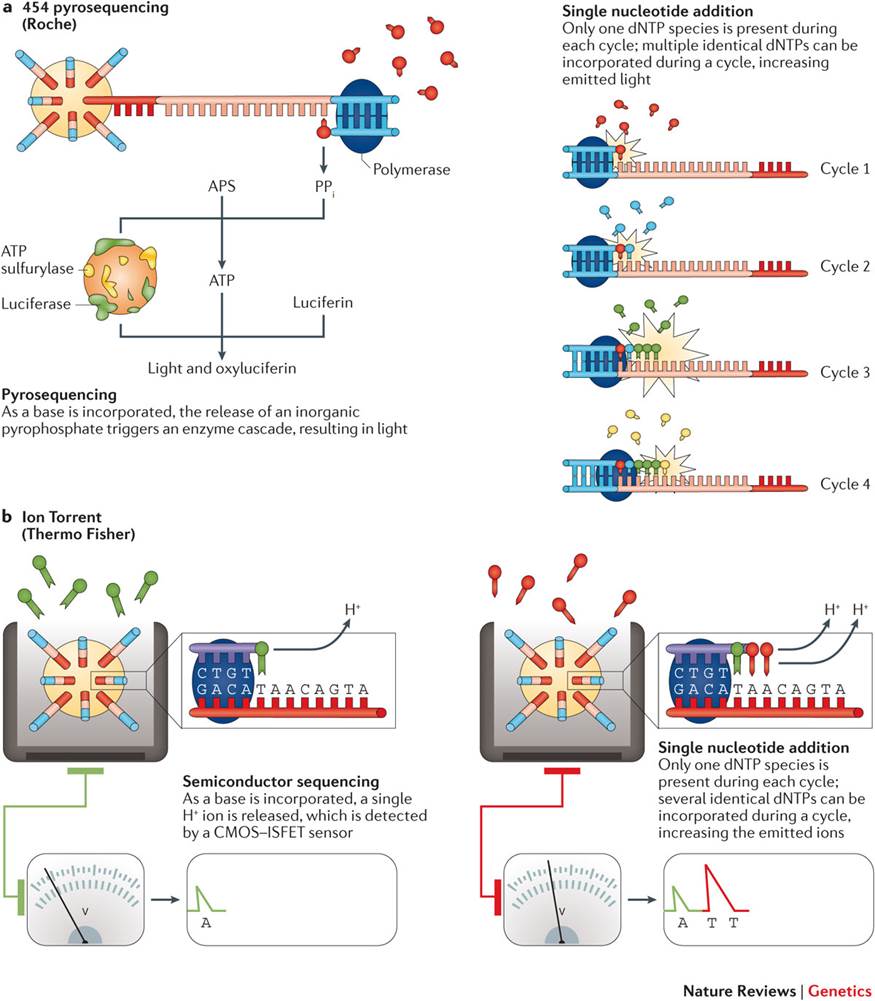 Sequencing by synthesis: single-nucleotide addition approaches
Sequencing by synthesis: single-nucleotide addition approaches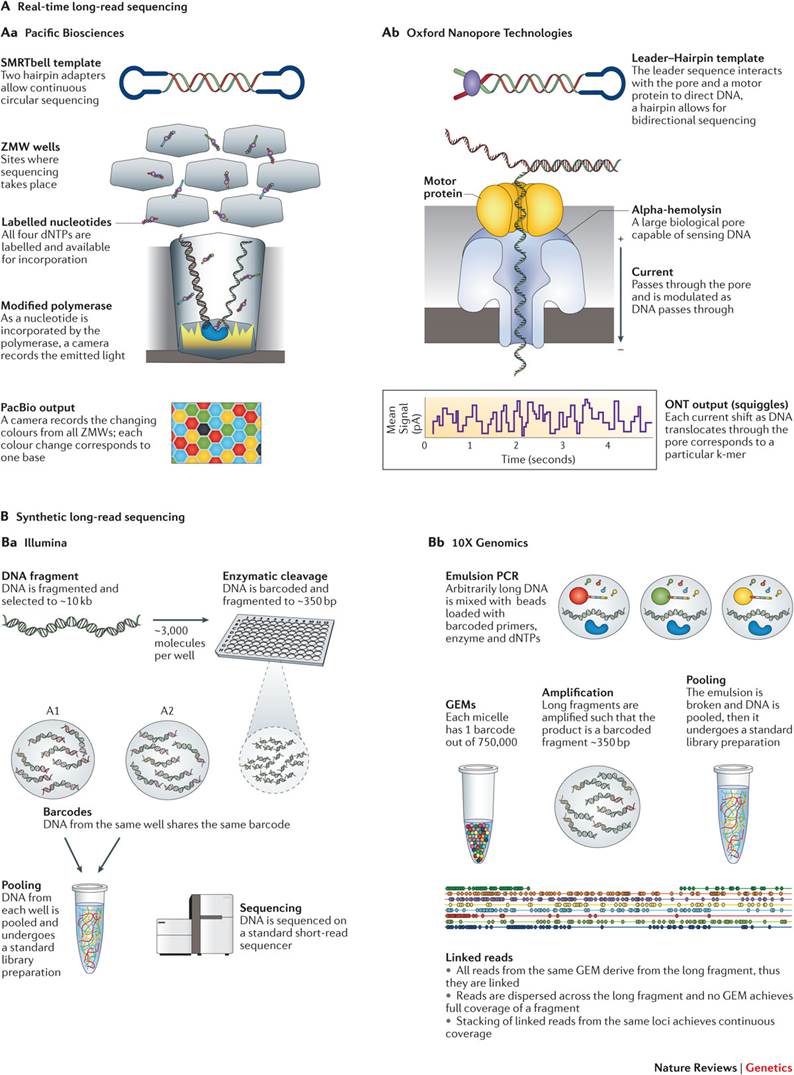 Sequencing by synthesis: single-nucleotide addition approaches
Sequencing by synthesis: single-nucleotide addition approaches