본 post는 국가생명연구자원정보센터(KOBIC) 주관 이화여자대학교 생명과학과 이상혁 교수님의 전사체 데이터 분석를 정리한 내용입니다.
Intro
RNA-seq과 microarray에 대한 전반적인 내용을 알아봅니다.
전사체 연구란?
- Omics: 어떤 대상을 모두 모아서 연구하는 학문
- Transcriptome 연구: 다양한 상황에서 RNA 전체를 대상으로 하는 연구
- 유전자 활동: 유전자 발현의 결과물(RNA, Protein 등)로 판단
유전자 발현
- where?
- tissue or organ profile. 장소에 따라 어떤 gene이 발현되느냐가 달라집니다.
- Under certain circumstances?
- temporal progress. 동일한 organ 내에서라도 시간에 따라 normal cell이 cancer cell로 변하면서 발현되는 gene의 종류와 양도 달라집니다.
- Gene expression and Gene regulation
- a “proxy” measure for transcription/translation events! RNA/protein의 발현 정도로 발현/조절을 파악할 수 있습니다.
- a “proxy” measure for transcription/translation events! RNA/protein의 발현 정도로 발현/조절을 파악할 수 있습니다.
Omics 연구의 장점
- survey of all components
- efficient for “biomarker discovery”
- unbiased view! “system biology”
Transcriptome 연구를 통해 해결할 수 있는 문제들
- compare two (or more) conditions to identify differentially expressed genes
- control vs. treatment
- disease vs. normal
- exploratory analysis
- what genes are expressed in response to drought stress?
- what gene expression changes occur during normal retinal development?
- diagnostic & prognostic tool development
- can we predict certain conditions (breast cancer vs. normal) 암 진단법을 만들 수 있습니다.
- can we identify patterns of gene expression that predict a patient’s response to treatment/drug? 치료 예후예측
사람의 몸에 침입한 antigen은 antigen-presenting cell에 의하여 우리 몸이 침입을 감지하고 대응할 수 있도록 알리는 역할을 합니다. Cytotoxic T-cell이 직접 antigen을 공격하는 동시에 helper T-cell이 B-cell로 하여금 antibody를 생성, 배출하도록 리드합니다. 또한 memory T-cell로 하여금 적응면역을 갖도록 합니다.
Applications of Gene Expression
- clustering
- gene function inference
- regulatory network
- molecular pathology
- classification
- diagnosis and prognosis
Transcriptome 연구 범위
- exporession profiling
- mRNA, miRNA, IncRNA, …
- alternative splicing
- differentially expressed genes (DEGs)
- gene set and pathway analysis
- precision medicine
- fusion gene analysis
- patient stratification
- cell type and composition inference
Transcriptome 연구 방법
- microarray: universal biochemistry platforms
![Post-Image]() microarray
microarray
https://www.edwith.org/transcriptome/lecture/1382678- micro(작은) + array(행렬): 2d 배열의 작은 hole에 물질을 넣고 분석할 수 있는 방식
- motivations: hundreds, thousands or even millions of individual experiments are conducted in parallel, with very few reagents. 즉, 동시에 20,000개~30,000개의 gene의 발현을 한 번에 알 수 있다는 점에서 효율적입니다.
- many different types
- DNA
- mRNA in cDNA
- proteins (antbodies)
- chemical compounds
- tissues
- carbohydrates
- principles
- extension of southern and northern blotting
- microarray는 gene에 specific한 sequence로 구성된 fixed probes가 hole마다 고정되어 있습니다. (hybridization-bases)
- normal vs. tumor의 mRNA를 각각 green, red dye를 붙인 뒤 1:1 비율로 microarray에 loading하면 normal vs. tumor에서 더 많이 발현되는 색상이 우세하게 나타납니다.
![Post-Image]() microarray principles
microarray principles
https://www.edwith.org/transcriptome/lecture/1382678
- Affymetrix expression arrays
- Affymetrix사에서 만든 expression arrays로, 25-mers 길이의 oligo로 구성되었습니다.
- Human Transcriptome Array 2.0은 285,000개 이상의 RNA 서열을 대상으로 발현양을 확인할 수 있습니다.
![Post-Image]() Affymetrix Expression Arrays
Affymetrix Expression Arrays
https://www.edwith.org/transcriptome/lecture/1382678
- RNA-Seq (Transcriptome Sequencing)
- the high throughput sequencing of cDNA using NGS technology
- the goal is to identify regions in the genome that are being transcribed in a sample
- strategies for reconstructing transcripts from RNA-Seq
- align-then-assemble: human reference genome에 align 한 뒤 gene별로 얼마나 붙는지 확인합니다. Quantification
- assemble-then-align: sequencing reads끼리 assembly 한 뒤 reference genome에 align 합니다. Novel RNAs를 확인할 수 있습니다.
![Post-Image]() RNA-Seq
RNA-Seq
https://www.edwith.org/transcriptome/lecture/1382678
- common analysis of RNA-Seq
- gene expression analysis
- differential expression analysis
- transcript discovery and annotation
- novel transcripts/genes
- noncoding RNAs: miRNAs, IncRNAs, …
- gene fusion detection (structural variants)
- alternative splicing event
- RNA-specific phenomena
- allele-specific expression: 부/모 어느 쪽에서 온 allele이 발현되는지 확인합니다.
- RNA editing
- Mutation discovery & prioritization
- advantages of RNA-Seq over microarray
- one sample reparation, various analysis
- RNA-Seq has higher resolution
- single-base pair resolution
- dynamic range of gene expression
- variatin of RNA-Seq
- single cell sequencing
- miRNA sequencing
- Long read sequencing
- challenges of RNA-Seq
- RNA is fragile compared to DNA (easily degraded) 샘플 변성이 매우 잘 일어납니다.
- Sample!!! purifu? quantity? quality?
- RNAs consist of small exons separated by introns
- the relative abundance of RNAs vary wildly
- RNAs come in a wide range of sizes
- RNA-Seq library enrichment strategies
- total RNA: all types RNAs가 sequencing 됩니다.
- rRNA reduction: rRNAs를 제거한 나머지 RNAs만 sequencing 합니다.
- PolyA selection: mRNA만 선별적으로 sequencing 합니다.
- cDNA capture: interesting genes의 mRNA만 선택하여 sequencing 합니다.
![Post-Image]() RNA-Seq library enrichment strategies
RNA-Seq library enrichment strategies
https://www.edwith.org/transcriptome/lecture/1382678
- stranded vs. unstranded
- 최근에는 대부분 stranded sequencing을 진행합니다.
![Post-Image]() stranded vs. unstranded
stranded vs. unstranded
https://www.edwith.org/transcriptome/lecture/1382678
- 최근에는 대부분 stranded sequencing을 진행합니다.
- RNA quality control
- RIN: RNA integrity number. 0(bad) ~ 10(good). RNA quality를 나타내는 수치입니다.
![Post-Image]() RIN
RIN
https://www.edwith.org/transcriptome/lecture/1382678
- RIN: RNA integrity number. 0(bad) ~ 10(good). RNA quality를 나타내는 수치입니다.
- replicates
- biological replicate: multiple isolations of cells showing the same phenotype, stage of other experimental condition. 즉, 시료의 양이 충분하다면 가급적 3개 이상으로 시료를 나누어 sequencing, analysis 하는 것을 권장합니다. 3개 중 1개 데이터에 이상이 있더라도 나머지 2개 데이터를 통계처리하여 올바른 결과를 도출할 수 있기 때문입니다.
- biological replicate: multiple isolations of cells showing the same phenotype, stage of other experimental condition. 즉, 시료의 양이 충분하다면 가급적 3개 이상으로 시료를 나누어 sequencing, analysis 하는 것을 권장합니다. 3개 중 1개 데이터에 이상이 있더라도 나머지 2개 데이터를 통계처리하여 올바른 결과를 도출할 수 있기 때문입니다.
Take Home Message
microarray 대비 RNA-Seq이 가지는 장점을 확인할 수 있었습니다. RNA-Seq에서 샘플의 quality가 매우 중요하고, 얻은 data로부터 gene expression level, gene fusion analysis, mutation discovery 등 다양한 결과를 얻을 수 있음을 확인 했습니다.
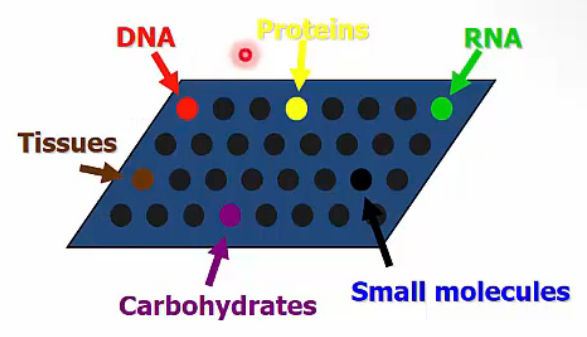 microarray
microarray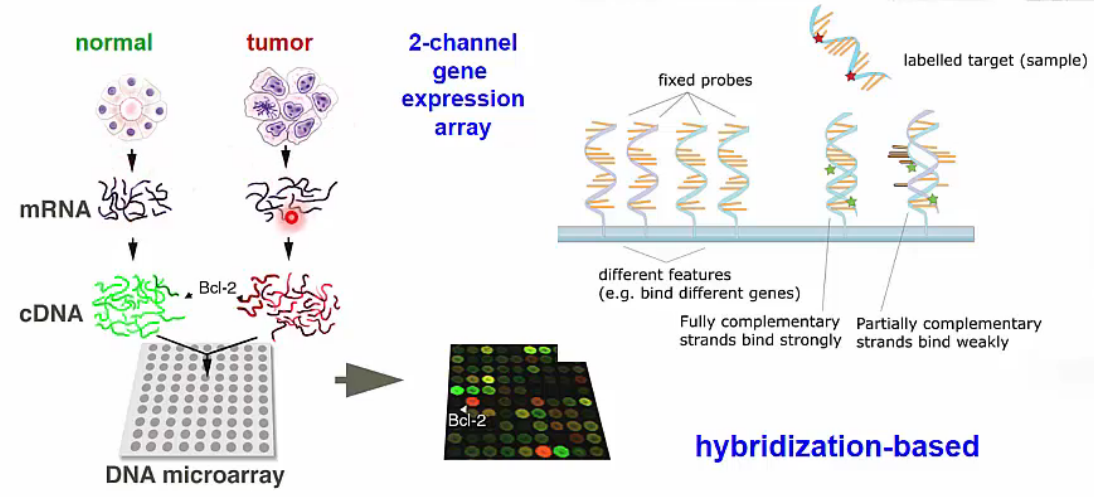 microarray principles
microarray principles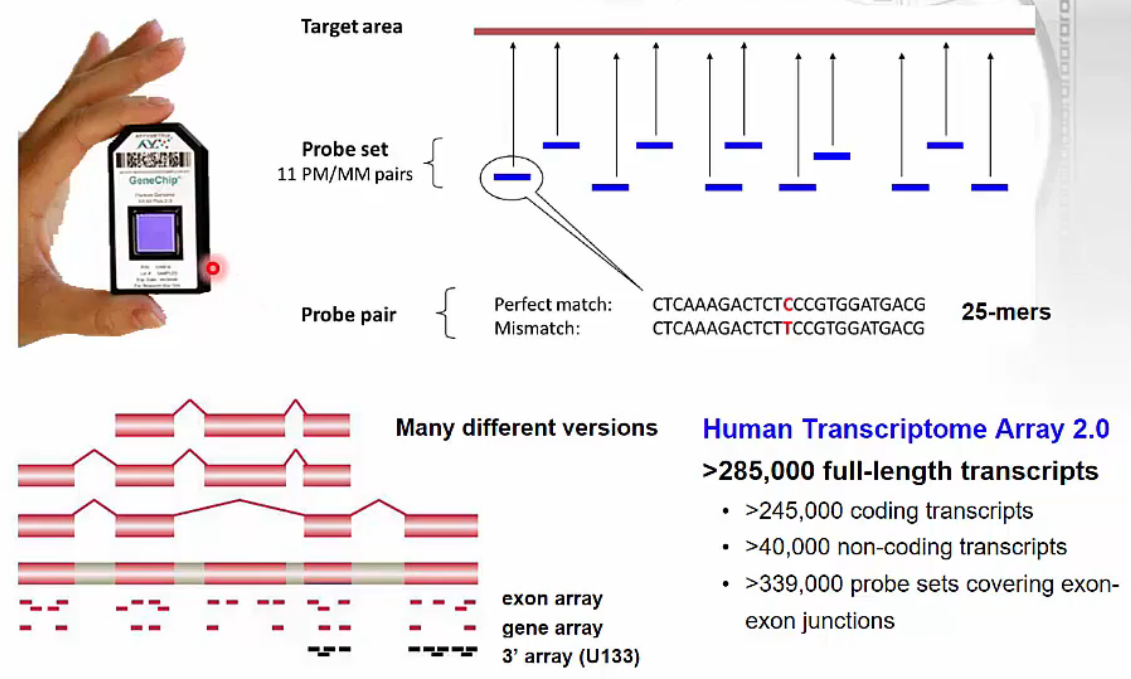 Affymetrix Expression Arrays
Affymetrix Expression Arrays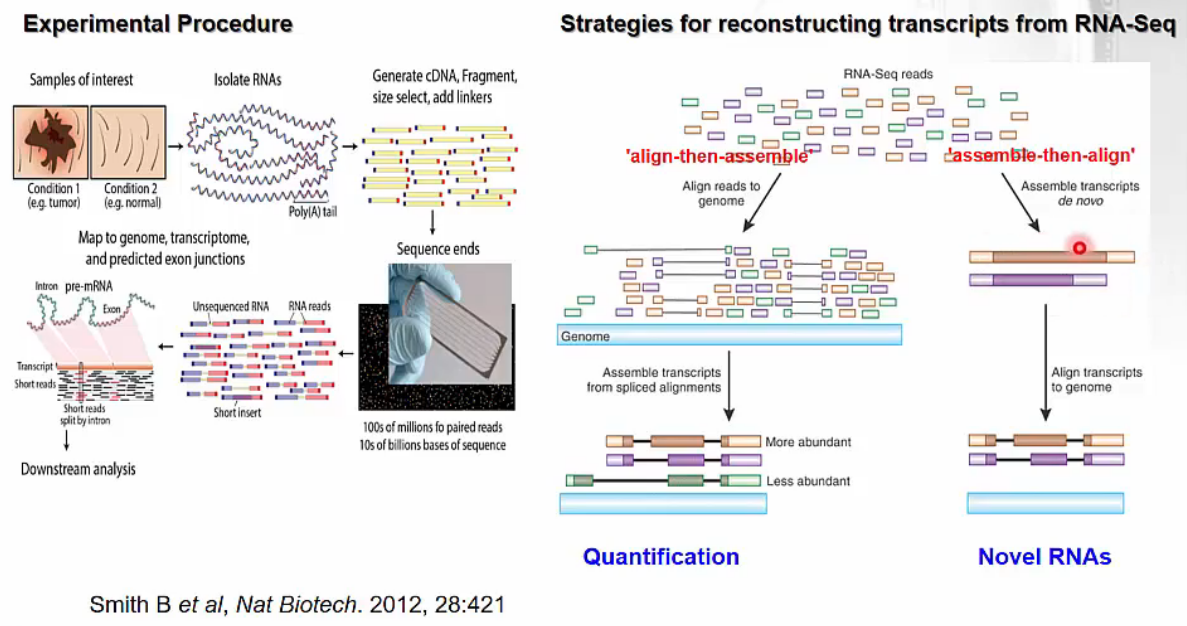 RNA-Seq
RNA-Seq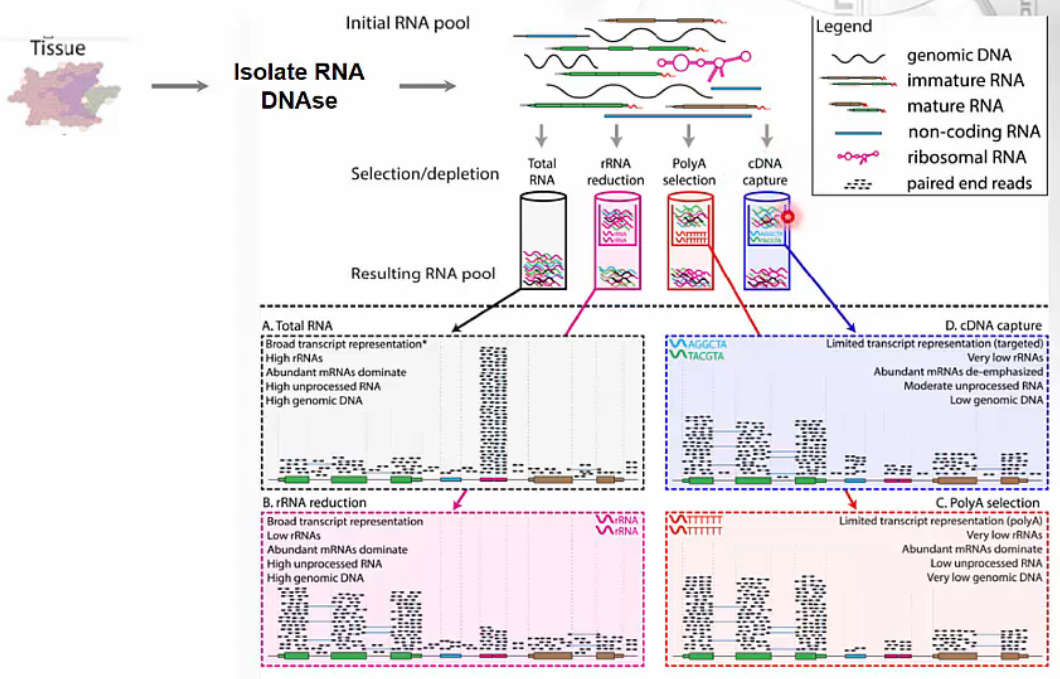 RNA-Seq library enrichment strategies
RNA-Seq library enrichment strategies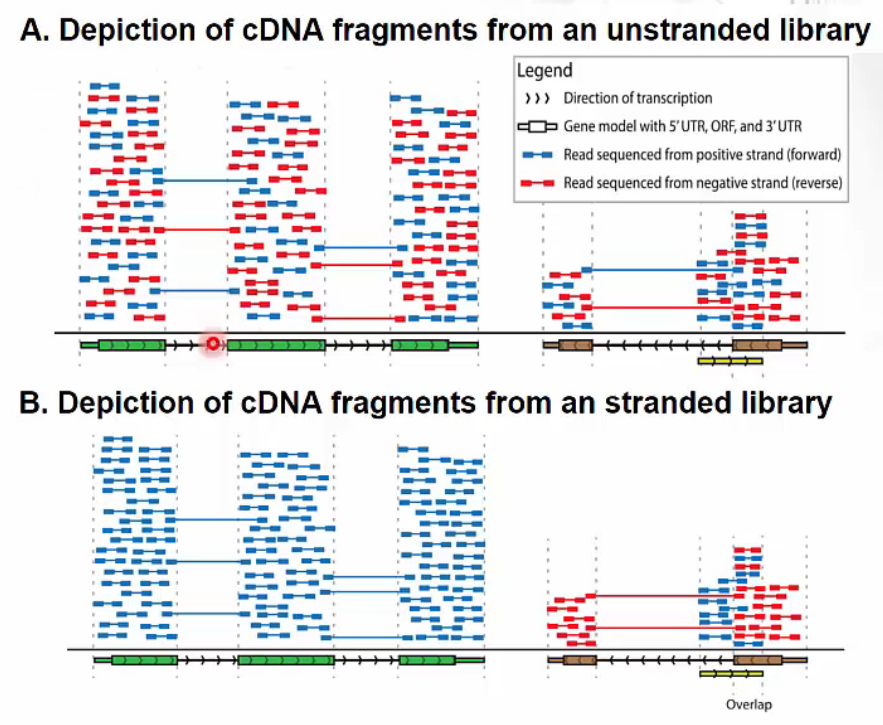 stranded vs. unstranded
stranded vs. unstranded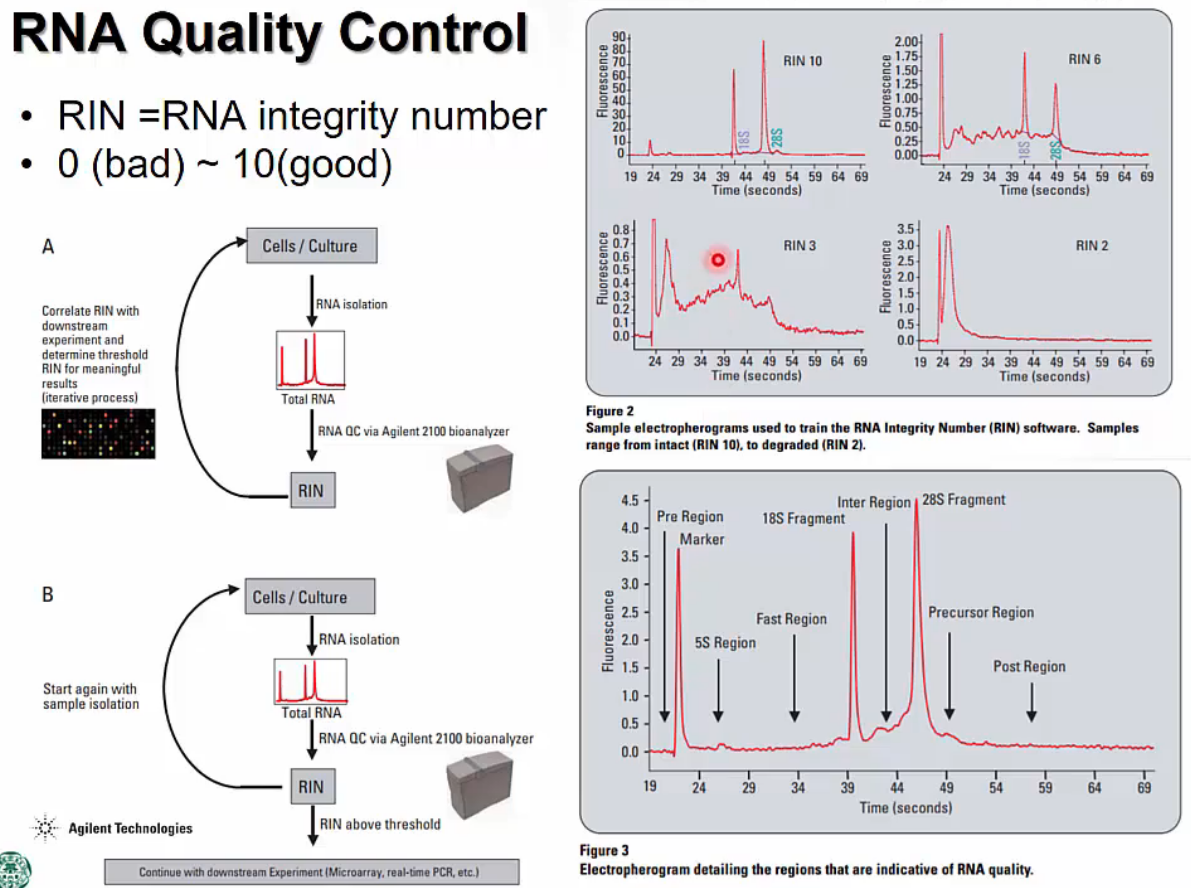 RIN
RIN