본 post는 국가생명연구자원정보센터(KOBIC) 주관 이화여자대학교 생명과학과 이상혁 교수님의 전사체 데이터 분석를 정리한 내용입니다.
Intro
RNA-seq data의 mapping, normalization, quantification에 대해 알아봅니다.
Quality Control
- Sequencing data를 얻으면 base quality(Q score)를 확인합니다.
- 사용 가능한 프로그램으로 FastQC, MultiQC, PRINSEQ, RSeQC 등이 있습니다.
Trimming
- Mapping 전 contaminated or low-quality reads를 제거하는 과정입니다.
- 사용 가능한 프로그램으로 Sickle, FASTX-Toolkit, Cutadapt, Trimmomatic 등이 있습니다.
 Reads Trimming
Reads Trimming
https://www.edwith.org/transcriptome/lecture/1382678
Sample Validation
- 다양한 이유로 sample이 서로 뒤바뀌는 경우가 발생할 수 있습니다. 이를 확인할 수 있는 다양한 방법이 있습니다.
- Paired sample shcek
- SNP concordance에 기반한 paired sample check 방법입니다.
- tool: NGSCheckMate, BAMixChecker
- Gender check
- X/Y chromosome 상에서 read depth나 allele frequency에 기반한 gender check 방법입니다.
- tool: SEXCMD
- Ethnicity inference
- 인종별로 나타나는 allele에 기반하여 ethnicity를 추정하는 방법으로 WGS or WES data를 대상으로 사용 가능합니다.
- tool: SeqSQC, EthSEQ, LASER 2.0
Mapping
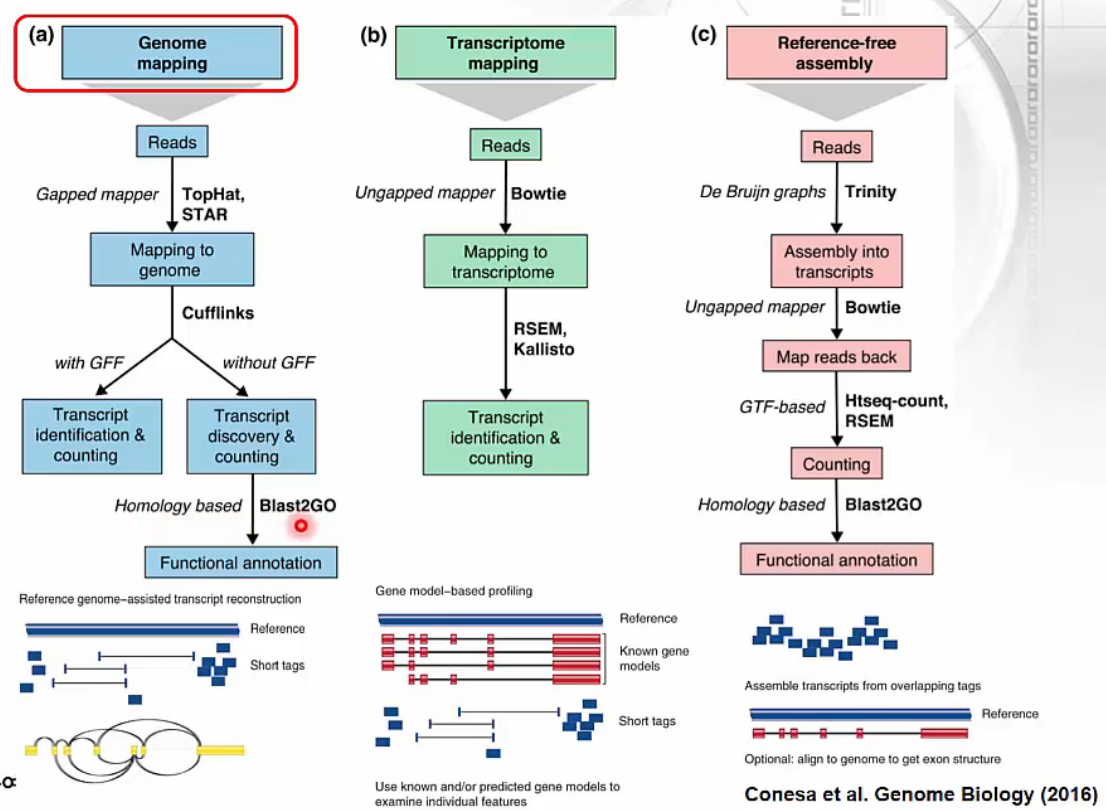 RNA-Seq Mapping
RNA-Seq Mapping
https://www.edwith.org/transcriptome/lecture/1382678
- 다양한 mapping 방법이 있습니다. 최근에는 genome에 mapping하는 방법이 주로 사용됩니다.
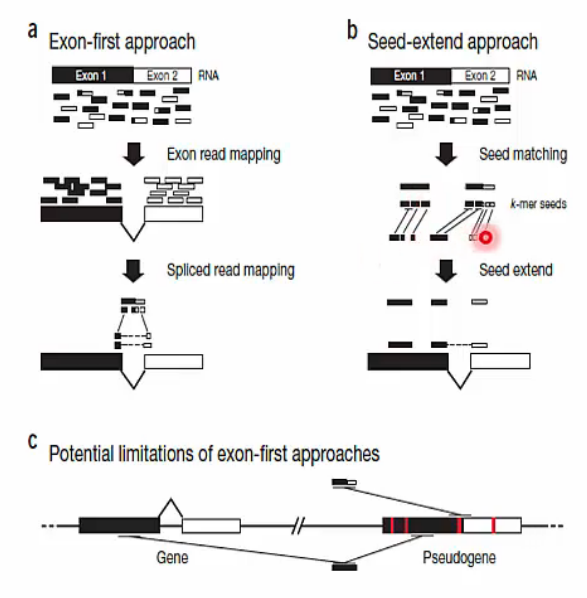 Strategies for gapped alignments
Strategies for gapped alignments
https://www.edwith.org/transcriptome/lecture/1382678
- RNA-Seq data를 genome에 mapping하는 방식은 크게 두 가지로 나눠 생각할 수 있습니다.
- Exon-first approach
- 이미 알려진 exon sequence에 read를 mapping하는 방식입니다.
- tool: MapSplice, SpliceMap, TopHat
- 하지만 gene과 pseudogene을 구분하지 못하고 모두 mapping 한다는 단점이 있습니다.
- Seed-extend methods
- N-mer의 seed reads가 어디에 matching 되는지 확인 후 확장시켜 나가는 방식입니다.
- tool: GSNAP, QPALMA
- Exon-first approach
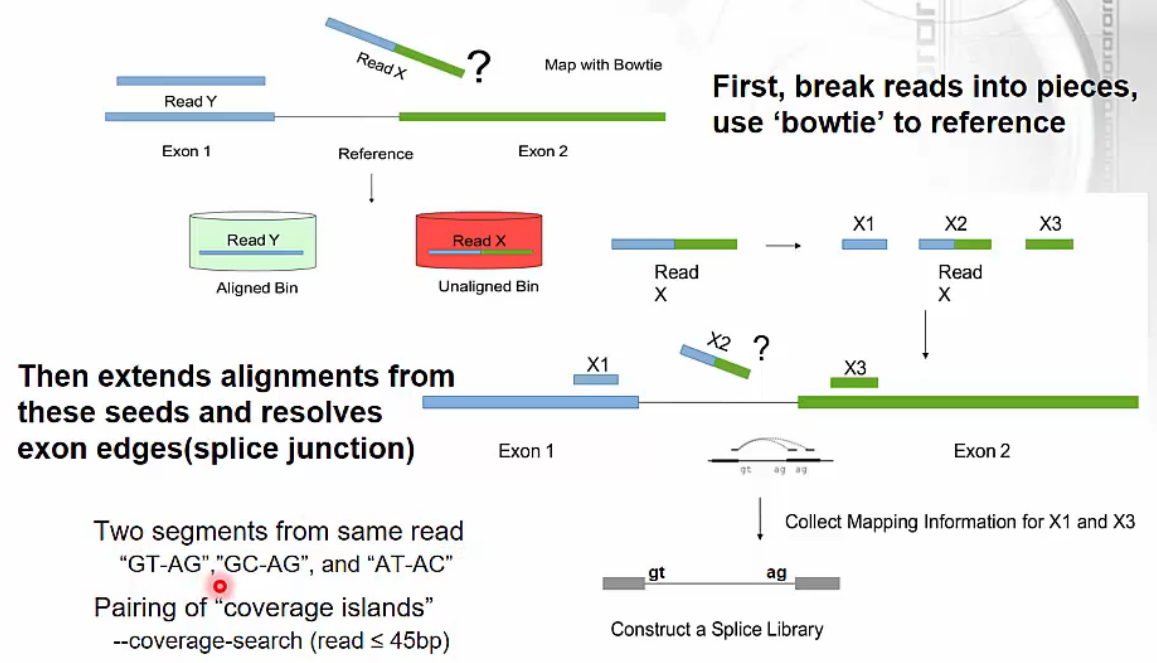 Tophat: Spliced Read Mapper
Tophat: Spliced Read Mapper
https://www.edwith.org/transcriptome/lecture/1382678
- Tophat - spliced read mapper
- Exon 내 mapping되는 reads를 먼저 선별합니다.
- 두 개의 exon에 걸쳐있는 reads는 각각 exon 영역에 맞는 영역으로 나눕니다. 이 때 canonical intron이 지니고 있는 특징적인 서열(intron의 시작과 끝 서열: GT-AG, GC-AG, AT-AC)을 활용합니다.
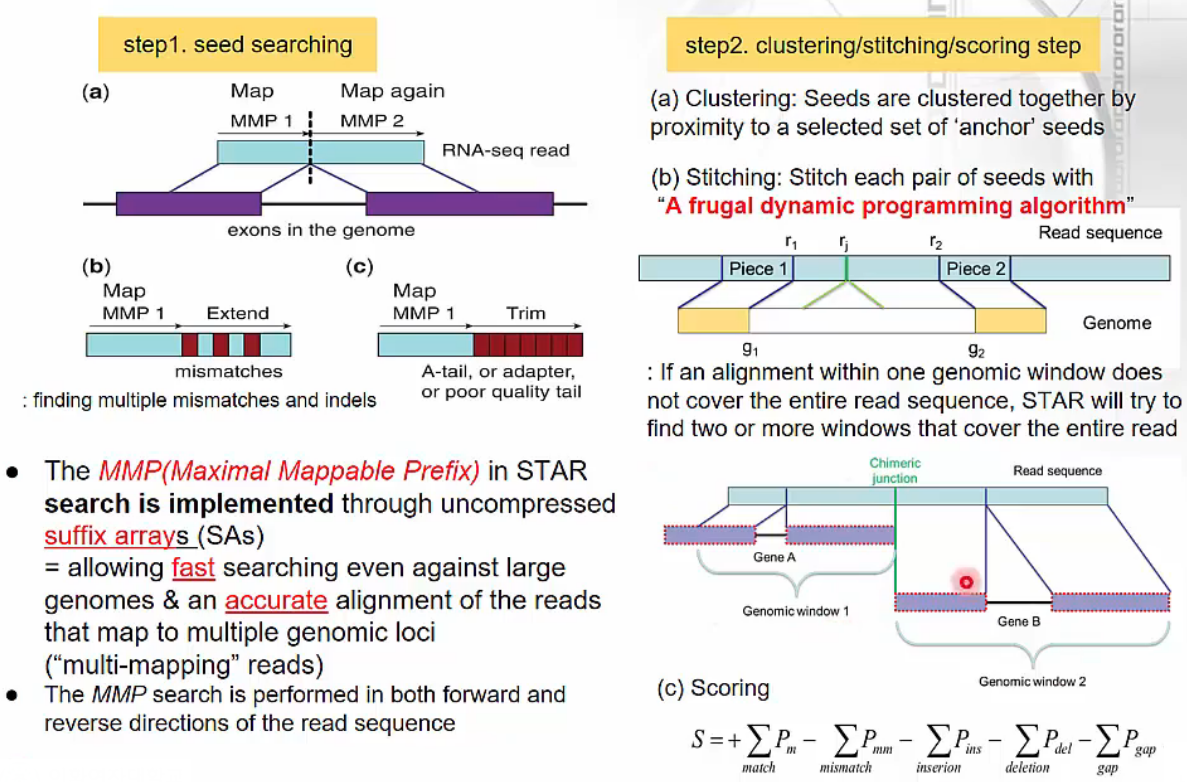 STAR: spliced transcripts alignment to a reference
STAR: spliced transcripts alignment to a reference
https://www.edwith.org/transcriptome/lecture/1382678
- STAR - spliced transcripts alignment to a reference
- step1. seed searching
- Suffiix array 알고리즘을 사용하여 MMP(Maximal Mappable Prefix)를 찾습니다.
- N-mer의 seed reads가 mapping되면 extend하면서 read를 연장합니다.
- setp2. clustering/stiching/scoring step
- clustering: seed reads를 모아 cluster를 만듭니다.
- stitching: frugal dynamic programming altorithm을 사용하여 각 pair of seeds를 연결합니다. 이 때 두 개 이상의 window를 사용하는데 scoring하여 가장 적합한 position을 찾습니다.
- step1. seed searching
- TopHat vs STAR
- TCGA RNA-Seq AML data를 사용하여 비교한 결과입니다.
- 동일한 data의 mapping에 TopHat2는 480분, STAR는 27분 소요됐습니다. STAR가 월등히 빠른 것을 알 수 있습니다.
- 하지만 정확도는 TopHat2가 약간 더 앞선 결과를 보였습니다.
- STAR를 사용하는 것이 더 효율적인 방법임을 생각해 볼 수 있습니다.
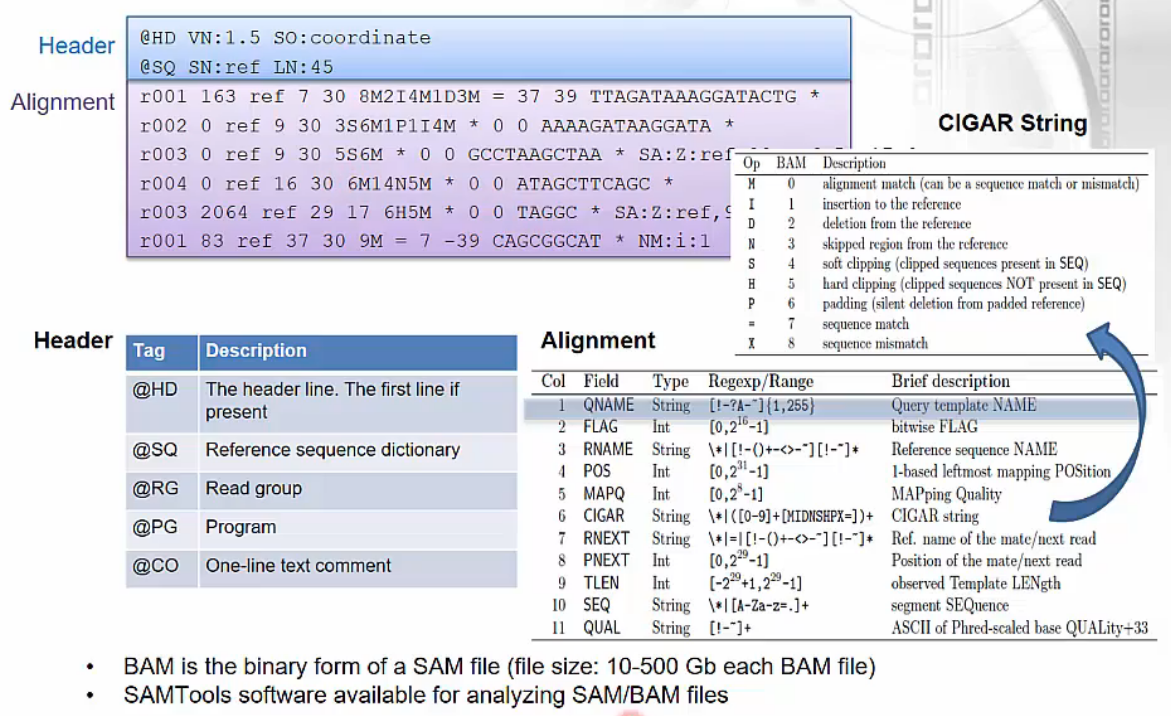 SAM/BAM
SAM/BAM
https://www.edwith.org/transcriptome/lecture/1382678
- Alignment Data Format (SAM/BAM)
- mapping 결과로 나오는 output 파일입니다.
- 6th column의 CIGAR string은 mapping 결과를 요약해서 보여주는데, RNA-Seq data에서 N은 intron 영역을 의미합니다.
- 예) 5M14N8M: exon 5bp + intron 14bp + exon 8bp
Normalization
- Normalization이 필요한 이유는, raw data가 mRNA의 concentration이 아닐 수 있기 때문입니다. 다양한 이유가 존재합니다.
- Sample preparation과 관련된 이슈가 있습니다.
- tissue contamination
- RNA degradation
- amplification efficiency
- reverse transcription efficiency
- microarrays와 관련된 이슈가 잇습니다.
- hybridization efficiency and specificity
- image segmentation
- signal quantificaion
- ‘background’ correction
- RNA-Seq과 관련된 이슈가 있습니다.
- uneven depth of coverage
- uncertainties in mapping and quantification
- Sample preparation과 관련된 이슈가 있습니다.
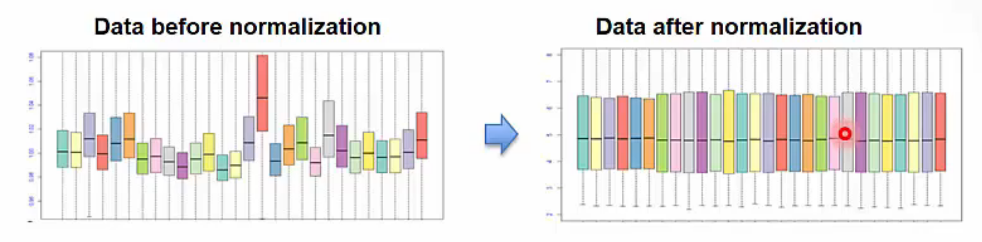 Normalization
Normalization
https://www.edwith.org/transcriptome/lecture/1382678
- Normalization은 데이터의 분포와 scale을 조정하여 데이터간 비교 가능하도록 만드는 과정입니다.
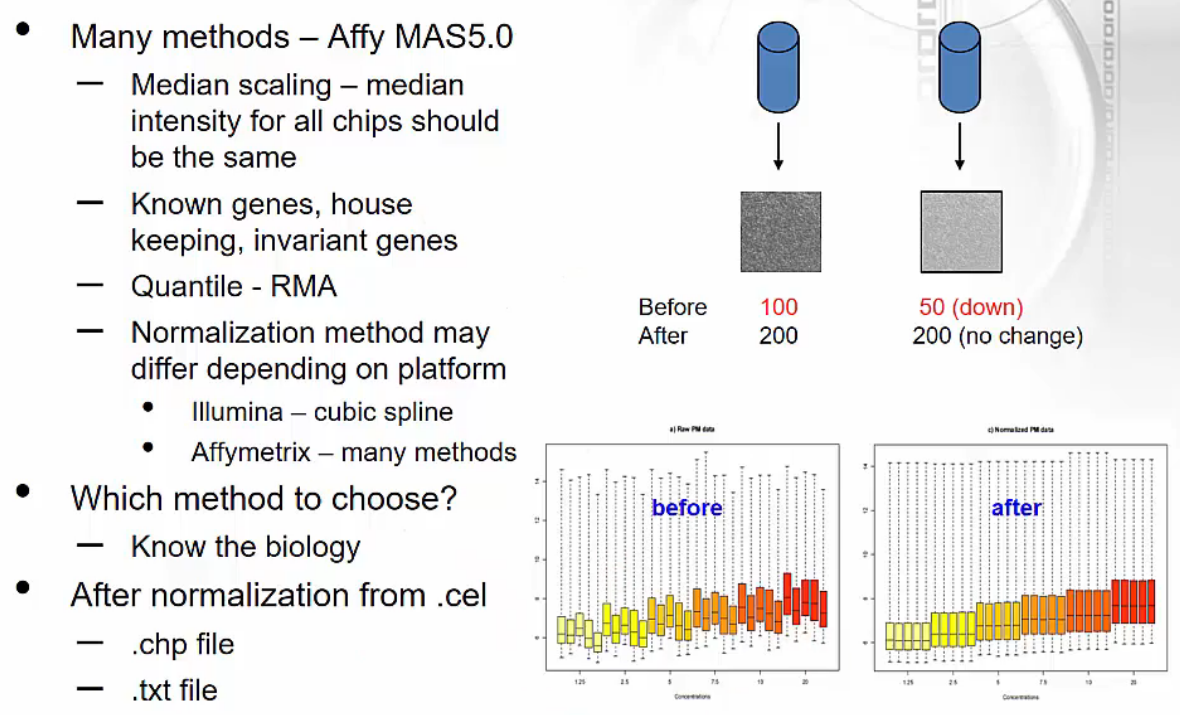 Normalization for microarray data
Normalization for microarray data
https://www.edwith.org/transcriptome/lecture/1382678
- Microarray data의 normalization은 여러 가지 방법이 존재합니다.
- 오른쪽 아래 그림을 보면 실제 duplicate였던 색상별 reads가 normalization 이후 동일한 수준으로 변경되었음을 확인할 수 있습니다.
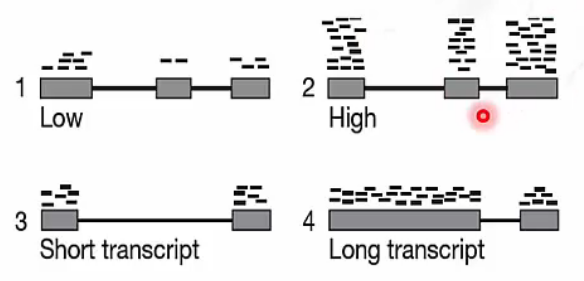 Normalization for RNA-Seq data
Normalization for RNA-Seq data
https://www.edwith.org/transcriptome/lecture/1382678
- RNA-Seq data를 normalization 할 때 두 가지 사항을 고려해야 합니다.
- 1-2: Sequencing depth가 높을수록(2) mapped reads도 증가합니다.
- 3-4: transcript length가 길수록(4) mapped reads도 증가합니다.
따라서 sequencing depth, transcript length에 대해 normalization이 필요합니다.
- RPKM
- Reads Per Kilobase of transcript per Million mapped reads
- reads 백만개 당 kilobase transcript에 mapping된 reads 수를 의미합니다.
- normalization order: depth first -> then length
- FPKM
- Fragments Per Kilobase …
- Paired-end sequencing을 했을 때 하나의 fragment에 대해 forward, reverse 두 번을 sequencing 하는데, 이를 한 개로 간주하고 계산한 결과입니다.
- TPM
- Transcripts Per Million
- normalization order: length first -> then depth
- TPM values can be compared between different samples directly because the sum of all TPMs in each sample art the same
- 지금은 TPM이 정석처럼 사용되고 있습니다.
Quanltification
 Read Counting Rules
Read Counting Rules
https://www.edwith.org/transcriptome/lecture/1382678
- gene 하나의 uniquely mapping되는 read가 있지만 복수 개의 genes에 걸쳐서 mapping되는 경우도 있습니다.
- 다음과 같은 방법으로 이러한 문제를 처리합니다.
- Estimating using unique reads (old)
- Uniquely mapped reads만 사용하는 방식입니다.
- 상대적으로 많은 reads를 버려야하는 단점이 잇습니다.
- tool: NEUMA
- Maximum Likelihood Estimation (MLE)
- low level expression은 정확하게 측정되지 않는 단점이 있습니다.
- tool: EMSAR, MISO, Cufflinks
- Expectation-Maximization
- 현재 가장 많이 사용하는 방식입니다.
- tool: RSEM, eXpress
- Estimating using unique reads (old)
RNA-Seq Analysis Pipeline
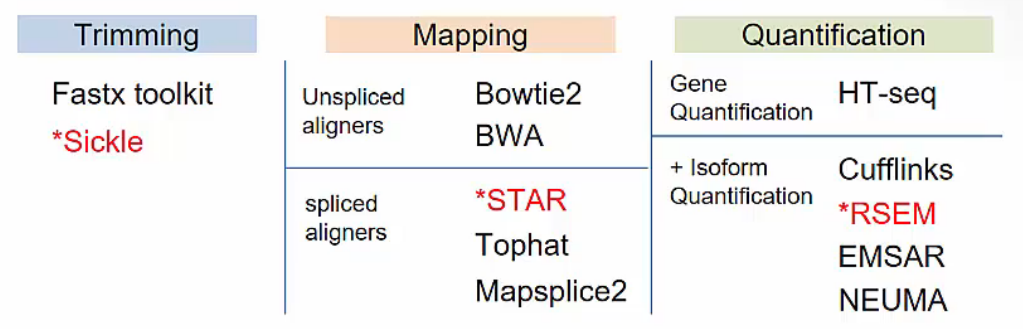 STAR-RSEM Anlysis Pipeline
STAR-RSEM Anlysis Pipeline
https://www.edwith.org/transcriptome/lecture/1382678
- Sickle / STAR / RSEM 을 사용한 RNA-Seq analysis pipeline 입니다.
- Trimming: Sickle
- Mapping STAR v2.6.0c
- Quantification: RSEM v1.3.0
- Differential expression analysis: R v3.6.0 (package: edgeR, preprocessCore, gplots, RColorbrewer)
- Fusion analysis: STARfusion v1.6.0
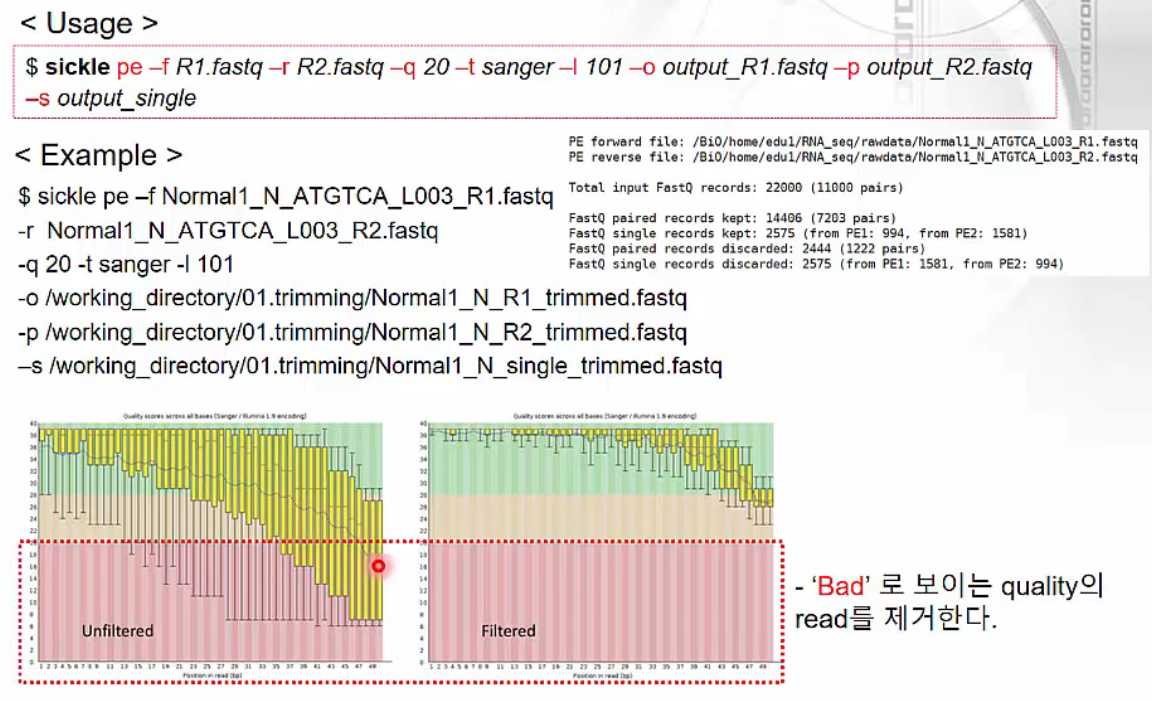 STAR-RSEM Anlysis Pipeline: trimming
STAR-RSEM Anlysis Pipeline: trimming
https://www.edwith.org/transcriptome/lecture/1382678
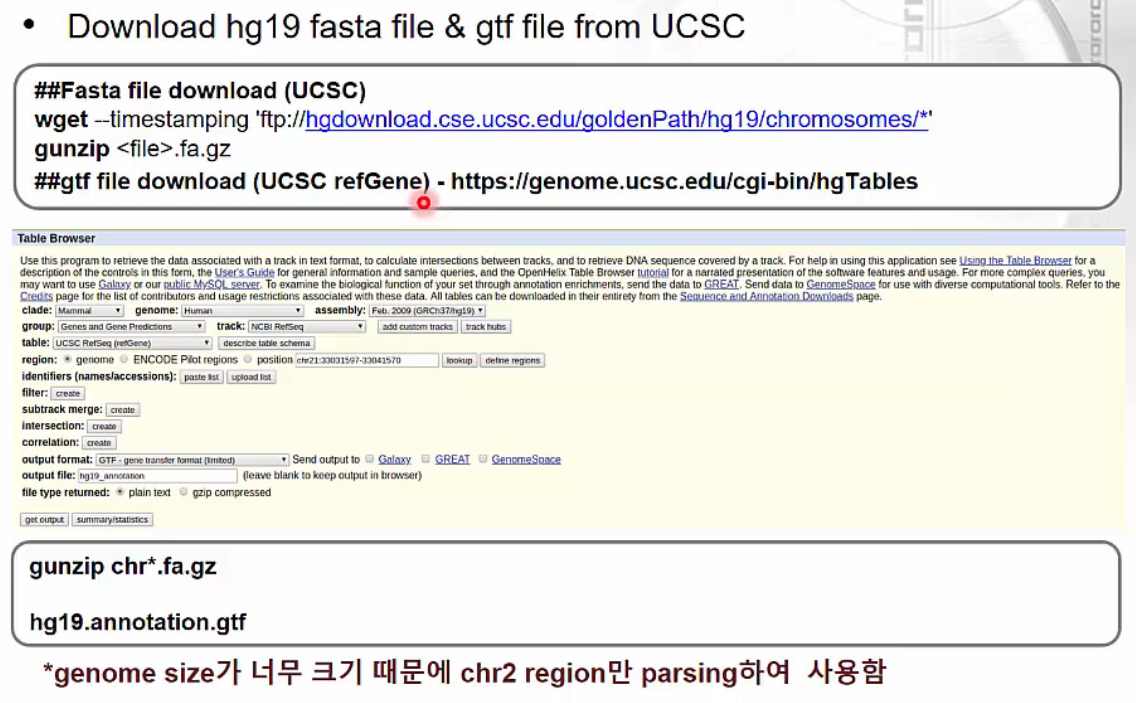 STAR-RSEM Anlysis Pipeline: preparing reference
STAR-RSEM Anlysis Pipeline: preparing reference
https://www.edwith.org/transcriptome/lecture/1382678
 STAR-RSEM Anlysis Pipeline: mapping & quantification
STAR-RSEM Anlysis Pipeline: mapping & quantification
https://www.edwith.org/transcriptome/lecture/1382678
 STAR-RSEM Anlysis Pipeline: mapping & quantification
STAR-RSEM Anlysis Pipeline: mapping & quantification
https://www.edwith.org/transcriptome/lecture/1382678
 STAR-RSEM Anlysis Pipeline: mapping & quantification
STAR-RSEM Anlysis Pipeline: mapping & quantification
https://www.edwith.org/transcriptome/lecture/1382678
Take Home Message
RNA-Seq data preprocessing 과정에 대해 배울 수 있었습니다. STAR-RSEM pipeline이 가장 많이 사용되고 있음을 알 수 있었습니다.