본 post는 국가생명연구자원정보센터(KOBIC) 주관 연세대학교 의과대학 김상우 교수님의 NGS 데이터 분석 기초 강의를 정리한 내용입니다.
Intro
NGS 최적 분석 파이프라인에 대해 알아봅니다.
Artigacts of variant calling
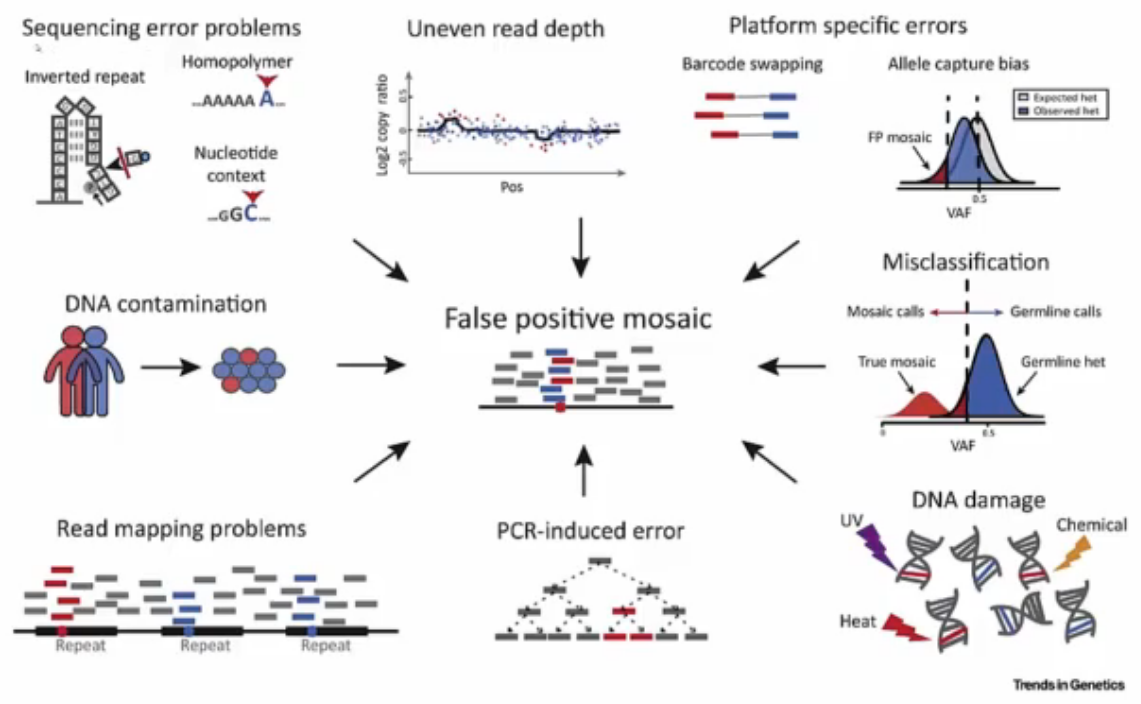 Artifacts of variant calling
Artifacts of variant calling
https://www.edwith.org/ngs-data-variation/joinLectures/356132
Variant calling 과정에 다양한 종류의 artifacts가 나타납니다. Sequencing error, uneven read depth, platform specific errors, misclassification, DNA damage, PCR-induced error, read mapping error, DNA contamination 등이 있습니다.
이 외에도 NGS data analysis 관련 다음과 같은 위험요소들이 있습니다.
Artifacts in sample handling
- Sample Swap
- 환자 A, B의 시료가 서로 뒤바뀌는 case로 생각보다 빈번하게 일어납니다.
- 해결1) Tumor, matched normal을 함께 가지고 있는 경우, 두 시료로부터 germline variants를 calling한 뒤 matrix를 그려보면 sample swap을 확인할 수 있습니다.
- 해결2) HYSYS, NGSCheckMate, BAMixChecker 등의 이미 개발된 tool을 활용하여 sample swap을 확인할 수 있습니다.
![Post-Image]() Check Sample Swap by Calling Germline Variants
Check Sample Swap by Calling Germline Variants
https://www.edwith.org/ngs-data-variation/joinLectures/356132 - Sample Contamination
- Human sample이 다른 종과 뒤섞인 case입니다.
- 해결) CONTEST 등의 이미 개발된 tool을 활용하여 sample contamination을 확인할 수 있습니다.
- DNA damage
- DNA는 보통 paraffin block에 보관하는데, 이 때 cytosine이 deamination 과정을 거쳐 uracil로 변하는 damage를 입게 됩니다.
- Paraffin block으로 오래 보관한 시료일수록 C>T false positive variant가 많이 나타남을 알 수 있습니다.
- 해결) 이미 개발된 tool을 활용하여 DNA damage false positive variant를 제거할 수 있습니다.
- Sample Swap
Artifacts in library preparation
- DNA damage by OxoG
- DNA capture 과정에서 OxoG(G>T, C>A variant) false positive variant가 발생합니다.
- 해결) 이미 개발된 tool을 활용하여 OxoG false positive variant를 제거할 수 있습니다.
- PCR-induced error
- PCR 과정에서 false positive variant가 발생합니다.
- DNA damage by OxoG
Low frequency variants
- Tumor와 normal이 섞여 있는 상태에서 low allele frequency variant calling은 매우 도전적인 과제입니다.
Platform specific errors
- Ion-Torrent: homopolymer 영역에서 정확한 base calling이 어렵습니다.
- 해결) ion-torrent에 특화된 analysis tool을 활용하여 해결할 수 있습니다.
Take Home Message
NGS variant calling은 단순한 pipeline 실행이 아닙니다. Human error, DNA damage, contamination, platform specific error 등 다양한 위험 요소가 존재합니다. 특히 환자의 genome analysis와 같은 실수가 용납되지 않는 analysis에서는 이러한 error를 고려한 pipeline 구성이 필수적입니다.
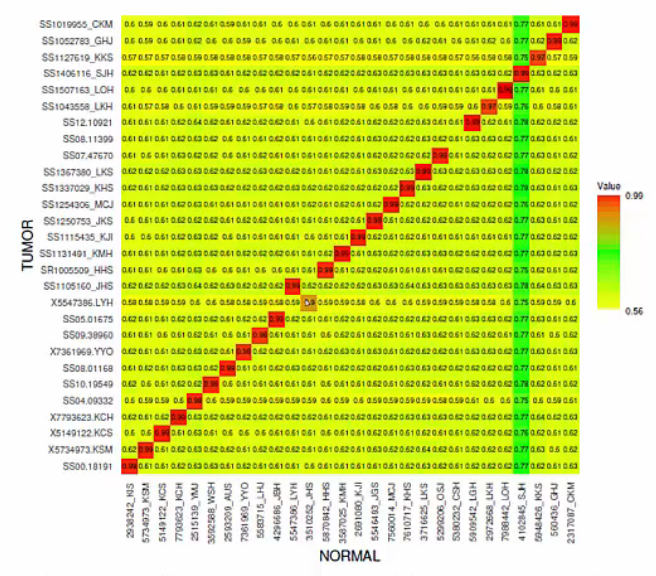 Check Sample Swap by Calling Germline Variants
Check Sample Swap by Calling Germline Variants